Chapitre 15 Qualité des données
Responsables du chapitre : Martijn Schuemie, Vojtech Huser & Clair Blacketer
La plupart des données utilisées pour la recherche observationnelle en soins de santé n’ont pas été collectées à des fins de recherche. Par exemple, les dossiers de santé électroniques (DSE) visent à capturer les informations nécessaires pour soutenir les soins aux patients, et les réclamations administratives sont collectées pour allouer les coûts aux payeurs. Beaucoup se sont interrogés sur l’opportunité d’utiliser de telles données pour la recherche clinique, avec Lei (1991) affirmant même que “Les données ne doivent être utilisées que pour l’objectif pour lequel elles ont été collectées.” La préoccupation est que, parce que les données n’ont pas été collectées pour la recherche que nous souhaitons mener, il n’est pas garanti qu’elles aient une qualité suffisante. Si la qualité des données est mauvaise (garbage in), alors la qualité des résultats de la recherche utilisant ces données doit également être mauvaise (garbage out). Un aspect important de la recherche observationnelle en soins de santé concerne donc l’évaluation de la qualité des données, visant à répondre à la question :
Les données ont-elles une qualité suffisante pour nos objectifs de recherche ?
Nous pouvons définir la qualité des données (DQ) comme (Roebuck 2012) :
L’état de complétude, de validité, de cohérence, d’actualité et de précision qui rend les données appropriées pour un usage spécifique.
Notez qu’il est peu probable que nos données soient parfaites, mais elles peuvent être suffisamment bonnes pour nos besoins.
La DQ ne peut pas être observée directement, mais une méthodologie a été développée pour l’évaluer. Deux types d’évaluations de DQ peuvent être distingués (Weiskopf and Weng 2013) : des évaluations pour évaluer la DQ en général, et des évaluations pour évaluer la DQ dans le contexte d’une étude spécifique.
Dans ce chapitre, nous examinerons d’abord les sources possibles de problèmes de DQ, puis nous discuterons de la théorie des évaluations de DQ générales et spécifiques à l’étude, suivie d’une description étape par étape de la manière dont ces évaluations peuvent être effectuées à l’aide des outils OHDSI.
15.1 Sources de problèmes de qualité des données
Il existe de nombreuses menaces à la qualité des données, commençant comme indiqué au Chapitre 14 lorsque le médecin enregistre ses réflexions. Dasu and Johnson (2003) distinguent les étapes suivantes dans le cycle de vie des données, recommandant d’intégrer la DQ à chaque étape. Ils appellent cela le continuum de la DQ :
- Collecte et intégration des données. Les problèmes possibles incluent la saisie manuelle faillible, les biais (par exemple, la surcodification dans les réclamations), la jonction erronée de tables dans un DSE, et le remplacement des valeurs manquantes par des valeurs par défaut.
- Stockage des données et partage des connaissances. Les problèmes potentiels sont le manque de documentation du modèle de données, et le manque de méta-données.
- Analyse des données. Les problèmes peuvent inclure des transformations de données incorrectes, une interprétation incorrecte des données, et l’utilisation de méthodologies inappropriées.
- Publication des données. Lors de la publication des données pour une utilisation ultérieure.
Souvent, les données que nous utilisons ont déjà été collectées et intégrées, il y a donc peu de choses que nous pouvons faire pour améliorer la première étape. Nous avons des moyens de vérifier la DQ produite par cette étape, comme il sera discuté dans les sections suivantes de ce chapitre.
De même, nous recevons souvent les données sous une forme spécifique, nous avons donc peu d’influence sur une partie de l’étape 2. Cependant, dans OHDSI nous convertissons toutes nos données observationnelles au modèle de données commun (CDM), et nous avons la maîtrise de ce processus. Certains ont exprimé des préoccupations selon lesquelles cette étape spécifique pourrait dégrader la DQ. Mais parce que nous contrôlons ce processus, nous pouvons mettre en place des garde-fous stricts pour préserver la DQ, comme discuté plus loin dans la Section 15.1.2. Plusieurs études (Defalco, Ryan, and Soledad Cepeda 2013; Makadia and Ryan 2014; Matcho et al. 2014; Voss, Ma, and Ryan 2015; Voss et al. 2015; G. Hripcsak et al. 2018) ont montré que, lorsqu’elle est correctement exécutée, peu ou pas d’erreurs sont introduites lors de la conversion au CDM. En fait, avoir un modèle de données bien documenté et partagé par une grande communauté facilite le stockage des données de manière non ambiguë et claire.
L’étape 3 (analyse des données) relève également de notre contrôle. Dans OHDSI, nous avons tendance à ne pas utiliser le terme DQ pour les problèmes de qualité lors de cette étape, mais plutôt les termes validité clinique, validité logicielle et validité méthodologique, qui sont longuement discutés dans les Chapitres 16, 17 et 18, respectivement. ## Qualité des données en général
Nous pouvons nous demander si nos données sont adaptées à l’objectif général de la recherche observationnelle. Michael G. Kahn et al. (2016) définissent cette qualité des données générique comme composée de trois éléments :
- Conformité : Les valeurs des données respectent-elles les normes et formats spécifiés ? Trois sous-types sont identifiés :
- Valeur : Les éléments de données enregistrés sont-ils en accord avec les formats spécifiés ? Par exemple, toutes les spécialités médicales des prestataires sont-elles des spécialités valides ?
- Relationnelle : Les données enregistrées sont-elles en accord avec les contraintes relationnelles spécifiées ? Par exemple, le PROVIDER_ID dans les données DRUG_EXPOSURE possède-t-il un enregistrement correspondant dans la table PROVIDER ?
- Calcul : Les calculs sur les données donnent-ils les résultats escomptés ? Par exemple, l’IMC calculé à partir de la taille et du poids est-il égal à l’IMC déclaré dans les données ?
- Complétude : Cela fait référence à la présence d’une variable particulière (par exemple, le poids mesuré dans le cabinet du médecin est-il enregistré ?) ainsi qu’à la présence de toutes les valeurs enregistrées pour les variables (par exemple, toutes les personnes ont-elles un sexe connu ?).
- Crédibilité : Les valeurs des données sont-elles crédibles ? Trois sous-types sont définis :
- Unicité : Par exemple, chaque PERSON_ID apparaît-il une seule fois dans la table PERSON ?
- Atemporelle : Les valeurs, distributions ou densités sont-elles en accord avec les valeurs attendues ? Par exemple, la prévalence du diabète indiquée par les données est-elle conforme à la prévalence connue ?
- Temporelle : Les changements de valeurs sont-ils conformes aux attentes ? Par exemple, les séquences de vaccination sont-elles en ligne avec les recommandations ?
Chaque composant peut être évalué de deux manières :
- Vérification se concentre sur les contraintes de données du modèle et des métadonnées, les hypothèses du système et la connaissance locale. Elle ne repose pas sur une référence externe. La caractéristique clé de la vérification est la capacité de déterminer les valeurs et distributions attendues en utilisant des ressources au sein de l’environnement local.
- Validation se concentre sur l’alignement des valeurs de données par rapport à des références externes pertinentes. Une source possible de référence externe peut être de combiner les résultats de plusieurs sites de données.
15.1.1 Vérifications de la qualité des données
Kahn introduit le terme data quality check (parfois appelé data quality rule) qui teste si les données sont conformes à une exigence donnée (par exemple, signaler un âge improbable de 141 ans pour un patient, potentiellement dû à une année de naissance incorrecte ou à un événement de décès manquant). Nous pouvons implémenter de telles vérifications dans des logiciels en créant des outils DQ automatisés. Un de ces outils est ACHILLES (Automated Characterization of Health Information at Large-scale Longitudinal Evidence Systems) (Huser et al. 2018). ACHILLES est un outil logiciel qui fournit une caractérisation et une visualisation d’une base de données conforme au CDM. À ce titre, il peut être utilisé pour évaluer la qualité des données dans un réseau de bases de données (Huser et al. 2016). ACHILLES est disponible en tant qu’outil autonome, et il est également intégré dans ATLAS en tant que fonction “Sources de données”.
ACHILLES pré-calculent plus de 170 analyses de caractérisation des données, chacune ayant un ID d’analyse et une courte description de l’analyse ; deux exemples sont “715: Distribution de DAYS_SUPPLY par DRUG_CONCEPT_ID” et “506: Distribution de l’âge au décès par sexe”. Les résultats de ces analyses sont stockés dans une base de données et peuvent être consultés par un visualiseur web ou par ATLAS.
Un autre outil créé par la communauté pour évaluer la qualité des données est le Data Quality Dashboard (DQD). Alors que ACHILLES lance des analyses de caractérisation pour fournir une compréhension visuelle globale d’une instance CDM, le DQD va table par table et champ par champ pour quantifier le nombre d’enregistrements dans un CDM qui ne sont pas conformes aux spécifications données. Au total, plus de 1 500 vérifications sont effectuées, chacune organisée selon le cadre de Kahn. Pour chaque vérification, le résultat est comparé à un seuil où un échec est considéré comme tout pourcentage de lignes violant dépassant cette valeur. Le tableau 15.1 montre quelques exemples de vérifications.
Tableau : (#tab:dqdExamples) Exemples de règles de qualité des données dans le Tableau de bord de la qualité des données.
| Fraction des lignes violées | Description de la vérification | Seuil | Statut |
|---|---|---|---|
| 0.34 | Une valeur oui ou non indiquant si le provider_id dans VISIT_OCCURRENCE est le type de données attendu selon les spécifications. | 0.05 | ÉCHEC |
| 0.99 | Le nombre et pourcentage de valeurs sources distinctes dans le champ measurement_source_value de la table MEASUREMENT mappées à 0. | 0.30 | ÉCHEC |
| 0.09 | Le nombre et pourcentage d’enregistrements ayant une valeur dans le champ drug_concept_id de la table DRUG_ERA qui ne sont pas conformes à la classe des ingrédients. | 0.10 | SUCCÈS |
| 0.02 | Le nombre et pourcentage d’enregistrements ayant une valeur dans le champ verbatim_end_date de DRUG_EXPOSURE qui survient avant la date dans le champ DRUG_EXPOSURE_START_DATE de la table DRUG_EXPOSURE. | 0.05 | SUCCÈS |
| 0.00 | Le nombre et pourcentage d’enregistrements ayant une valeur en double dans le champ procedure_occurrence_id de PROCEDURE_OCCURRENCE. | 0.00 | SUCCÈS |
Dans l’outil, les vérifications sont organisées de plusieurs manières, l’une d’elles étant en vérifications de niveau table, champ et concept. Les vérifications au niveau table sont effectuées à un niveau élevé dans le CDM, par exemple pour déterminer si toutes les tables requises sont présentes. Les vérifications au niveau champ sont effectuées de manière à évaluer chaque champ dans chaque table pour leur conformité aux spécifications CDM. Cela inclut de s’assurer que toutes les clés primaires sont vraiment uniques et que tous les champs de concepts standards contiennent des identifiants de concepts dans le domaine correct, parmi beaucoup d’autres. Les vérifications au niveau concept vont un peu plus loin pour examiner les identifiants de concepts individuels. Beaucoup d’entre elles appartiennent à la catégorie de la plausibilité du cadre de Kahn, comme s’assurer que les concepts spécifiques au genre ne sont pas attribués à des personnes du mauvais genre (par exemple, le cancer de la prostate chez une patiente).
ACHILLES et DQD sont exécutés contre les données dans le CDM. Les problèmes de qualité des données identifiés de cette manière peuvent être dus à la conversion au CDM, mais peuvent également refléter des problèmes de qualité des données déjà présents dans les données sources. Si la conversion est en cause, il est généralement sous notre contrôle de remédier au problème, mais si les données sous-jacentes sont en cause, la seule solution peut être de supprimer les enregistrements problématiques.
15.1.2 Tests unitaires ETL
En plus des vérifications de la qualité des données à un niveau élevé, des vérifications des données au niveau individuel doivent être effectuées. Le processus ETL (Extract-Transform-Load) par lequel les données sont converties en CDM est souvent assez complexe, et avec cette complexité vient le danger de commettre des erreurs qui peuvent passer inaperçues. De plus, au fil du temps, le modèle de données source peut changer, ou le CDM peut être mis à jour, rendant nécessaire la modification du processus ETL. Les changements à un processus aussi complexe qu’un ETL peuvent avoir des conséquences inattendues, nécessitant que tous les aspects de l’ETL soient réexaminés et revus.
Pour s’assurer que l’ETL fait ce qu’il est censé faire, et continue à le faire, il est fortement recommandé de construire un ensemble de tests unitaires. Un test unitaire est un petit morceau de code qui vérifie automatiquement un seul aspect. L’outil Rabbit-in-a-Hat décrit dans le chapitre 6 peut créer un cadre de tests unitaires qui facilite l’écriture de tels tests unitaires. Ce cadre est un ensemble de fonctions R créées spécifiquement pour la base de données source et la version CDM cible de l’ETL. Certaines de ces fonctions servent à créer des entrées de données fictives conformes au schéma des données sources, tandis que d’autres fonctions peuvent être utilisées pour spécifier des attentes sur les données au format CDM. Voici un exemple de test unitaire :
source("Framework.R")
declareTest(101, "Person gender mappings")
add_enrollment(member_id = "M000000102", gender_of_member = "male")
add_enrollment(member_id = "M000000103", gender_of_member = "female")
expect_person(PERSON_ID = 102, GENDER_CONCEPT_ID = 8507)
expect_person(PERSON_ID = 103, GENDER_CONCEPT_ID = 8532)Dans cet exemple, le cadre généré par Rabbit-in-a-Hat est chargé, important les fonctions utilisées dans le reste du code. Nous déclarons ensuite que nous commencerons à tester les mappages du sexe des personnes. Le schéma source a une table ENROLLMENT, et nous utilisons la fonction add_enrollment créée par Rabbit-in-a-Hat pour créer deux entrées avec des valeurs différentes pour les champs MEMBER_ID et GENDER_OF_MEMBER. Enfin, nous spécifions l’attente qu’après l’ETL, deux entrées doivent exister dans la table PERSON avec diverses valeurs attendues.
Notez que la table ENROLLMENT a de nombreux autres champs, mais nous ne nous soucions pas beaucoup des valeurs de ces autres champs dans le cadre de ce test. Cependant, laisser ces valeurs (par exemple, la date de naissance) vides pourrait amener l’ETL à ignorer l’enregistrement ou à générer une erreur. Pour surmonter ce problème tout en gardant le code de test facile à lire, la fonction add_enrollment assignera des valeurs par défaut (les valeurs les plus communes observées dans le rapport de scan White Rabbit) aux champs de valeurs non spécifiés par l’utilisateur.
Des tests unitaires similaires peuvent être créés pour toute autre logique dans un ETL, aboutissant généralement à des centaines de tests. Lorsque nous avons terminé de définir le test, nous pouvons utiliser le cadre pour générer deux ensembles d’instructions SQL, l’un pour créer les données sources fictives et l’autre pour créer les tests sur les données ETL-isées :
insertSql <- generateInsertSql(databaseSchema = "source_schema")
testSql <- generateTestSql(databaseSchema = "cdm_test_schema")Le processus global est illustré dans la figure 15.1.
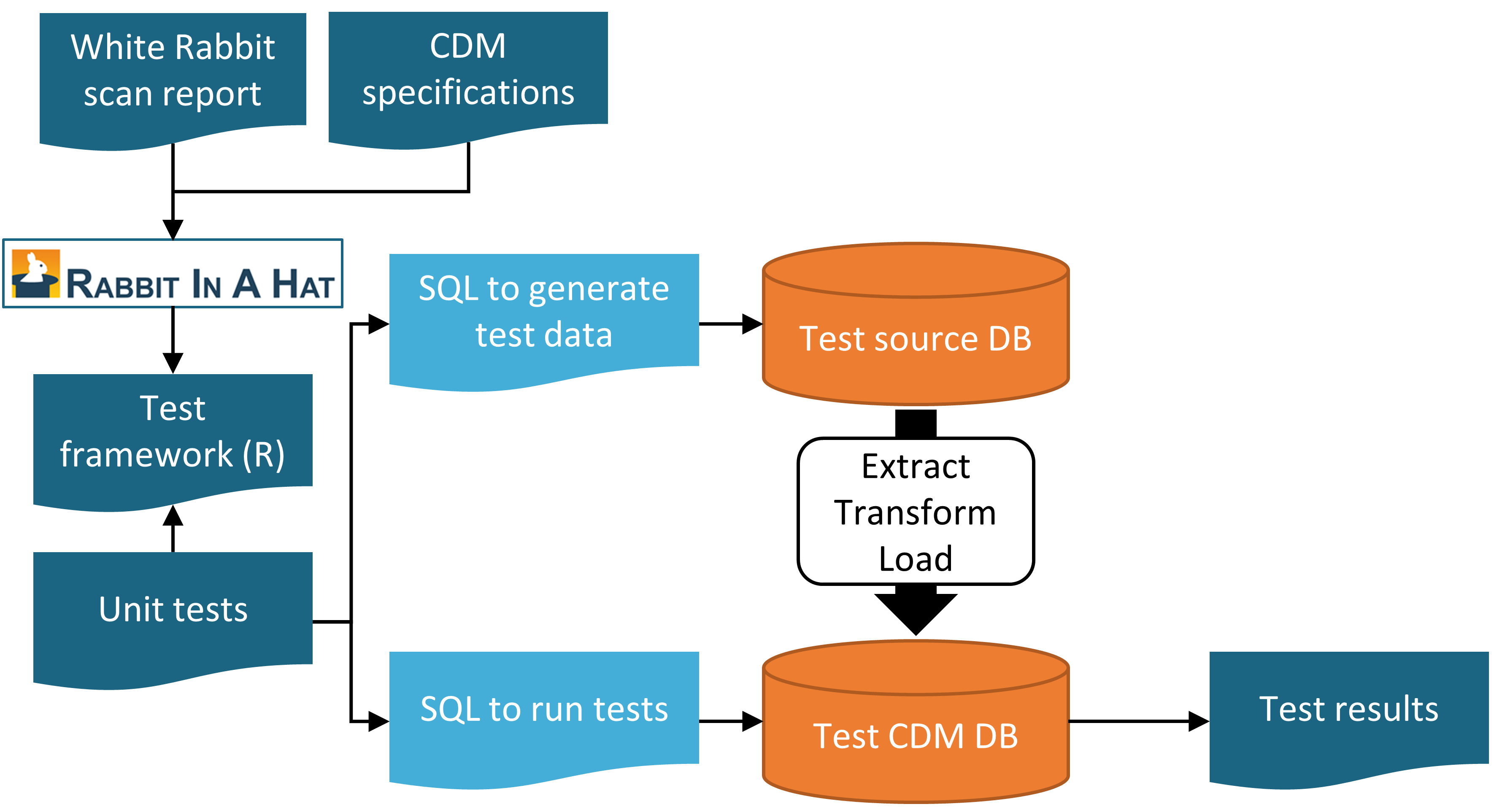
Figure 15.1: Unit testing an ETL (Extract-Transform-Load) process using the Rabbit-in-a-Hat testing framework.
Le test SQL retourne un tableau qui ressemblera à celui du tableau 15.2. Dans ce tableau, nous voyons que nous avons réussi les deux tests que nous avions définis plus tôt.
Tableau : (#tab:exampleTestResults) Exemples de résultats de tests unitaires ETL.
| ID | Description | Statut |
|---|---|---|
| 101 | Person gender mappings | SUCCÈS |
| 101 | Person gender mappings | SUCCÈS |
La puissance de ces tests unitaires est que nous pouvons facilement les relancer à chaque fois que le processus ETL est modifié.
15.2 Vérifications Spécifiques à l’Étude
Le chapitre s’est jusqu’ici concentré sur des vérifications générales de la qualité des données (DQ). Ces vérifications devraient être effectuées avant d’utiliser les données pour la recherche. Comme ces vérifications sont effectuées indépendamment des questions de recherche, nous recommandons d’effectuer des évaluations de DQ spécifiques à l’étude.
Certaines de ces évaluations peuvent prendre la forme de règles de DQ spécifiquement pertinentes pour l’étude. Par exemple, on peut souhaiter imposer une règle selon laquelle au moins 90% des enregistrements pour notre exposition d’intérêt précisent la durée de l’exposition.
Une évaluation standard consiste à passer en revue les concepts les plus pertinents pour l’étude dans ACHILLES, par exemple ceux spécifiés dans les définitions de cohortes de l’étude. Des changements soudains dans le temps du taux d’observation d’un code peuvent indiquer des problèmes de DQ. Quelques exemples seront discutés plus loin dans ce chapitre.
Une autre évaluation consiste à examiner la prévalence et les changements de prévalence au fil du temps des cohortes résultantes générées en utilisant les définitions de cohortes développées pour l’étude, et à voir si elles sont en accord avec les attentes basées sur des connaissances cliniques externes. Par exemple, l’exposition à un nouveau médicament devrait être absente avant son introduction sur le marché, et augmentera probablement avec le temps après son introduction. De même, la prévalence des résultats devrait être conforme à ce qui est connu de la prévalence de la condition dans la population. Si une étude est exécutée sur un réseau de bases de données, nous pouvons comparer la prévalence des cohortes entre les bases de données. Si une cohorte est très répandue dans une base de données, mais manque dans une autre base de données, il pourrait y avoir un problème de DQ. Notez que cette évaluation chevauche quelque peu la notion de validité clinique, comme discuté dans le Chapitre 16; nous pouvons trouver une prévalence inattendue dans certaines bases de données non pas à cause de problèmes de DQ, mais parce que notre définition de cohorte ne capture pas véritablement les états de santé qui nous intéressent, ou parce que ces états de santé varient légitimement entre les bases de données qui capturent différentes populations de patients.
15.2.1 Vérification des Mappings
Une source possible d’erreur qui relève entièrement de notre contrôle est le mapping des codes sources aux Concepts Standards. Les mappings dans le Vocabulaire sont méticuleusement élaborés, et les erreurs dans le mapping notées par les membres de la communauté sont reportées dans le traqueur de problèmes du Vocabulaire57 et corrigées dans les versions futures. Néanmoins, il est impossible de vérifier complètement tous les mappings manuellement, et des erreurs subsistent probablement. Lors de l’exécution d’une étude, nous recommandons donc de passer en revue les mappings pour les concepts les plus pertinents pour l’étude. Heureusement, cela peut être réalisé assez facilement car dans le CDM nous stockons non seulement les Concepts Standards, mais aussi les codes sources. Nous pouvons passer en revue à la fois les codes sources qui se mappent aux concepts utilisés dans l’étude, ainsi que ceux qui ne se mappent pas.
Une façon de passer en revue les codes sources qui se mappent est d’utiliser la fonction checkCohortSourceCodes dans le package R MethodEvaluation. Cette fonction utilise une définition de cohorte telle que créée par ATLAS comme entrée, et pour chaque ensemble de concepts utilisé dans la définition de cohorte, elle vérifie quels codes sources se mappent aux concepts de l’ensemble. Elle calcule également la prévalence de ces codes dans le temps pour aider à identifier les problèmes temporels associés à des codes sources spécifiques. La sortie d’exemple dans la Figure 15.2 montre une décomposition partielle d’un ensemble de concepts appelé “Trouble dépressif.” Le concept le plus prévalent dans cet ensemble de concepts dans la base de données d’intérêt est le concept 440383 (“Trouble dépressif”). Nous voyons que trois codes sources dans la base de données se mappent à ce concept : le code CIM-9 3.11, et les codes CIM-10 F32.8 et F32.89. À gauche, nous voyons que le concept dans son ensemble montre d’abord une augmentation graduelle dans le temps, mais ensuite montre une chute brutale. Si nous regardons les codes individuels, nous voyons que cette chute peut être expliquée par le fait que le code CIM-9 cesse d’être utilisé au moment de la chute. Bien que ce soit le même moment où les codes CIM-10 commencent à être utilisés, la prévalence combinée des codes CIM-10 est bien plus faible que celle du code CIM-9. Cet exemple spécifique était dû au fait que le code CIM-10 F32.9 (“Trouble dépressif majeur, épisode unique, non spécifié”) aurait également dû se mapper au concept. Ce problème a depuis été résolu dans le Vocabulaire.
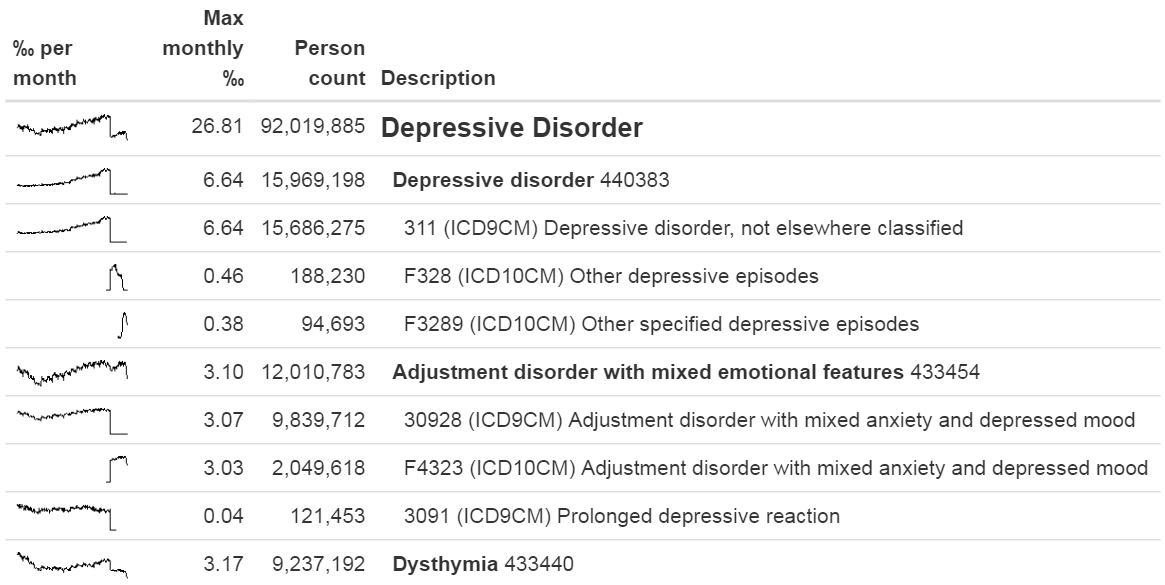
Figure 15.2: Example output of the checkCohortSourceCodes function.
Bien que l’exemple précédent démontre une découverte fortuite d’un code source qui n’était pas mappé, en général l’identification des mappings manquants est plus difficile que la vérification des mappings présents. Cela nécessite de savoir quels codes sources devraient se mapper mais ne le font pas. Une façon semi-automatisée de réaliser cette évaluation est d’utiliser la fonction findOrphanSourceCodes dans le package R MethodEvaluation. Cette fonction permet de rechercher le vocabulaire pour des codes sources en utilisant une recherche de texte simple, et elle vérifie si ces codes sources se mappent à un concept spécifique ou à l’un des descendants de ce concept. L’ensemble de codes sources résultant est ensuite restreint à uniquement ceux qui apparaissent dans la base de données CDM en question. Par exemple, dans une étude le concept “Trouble gangreneux” (439928) et tous ses descendants ont été utilisés pour trouver toutes les occurrences de gangrène. Pour évaluer si cela inclut vraiment tous les codes sources indiquant la gangrène, plusieurs termes (par exemple, “gangrène”) ont été utilisés pour rechercher les descriptions dans les tables CONCEPT et SOURCE_TO_CONCEPT_MAP pour identifier des codes sources. Une recherche automatisée est alors utilisée pour évaluer si chaque code source de gangrène apparaissant dans les données se mappe effectivement directement ou indirectement (via l’ascendance) au concept “Trouble gangreneux.” Le résultat de cette évaluation est montré dans la Figure 15.3, révélant que le code CIM-10 J85.0 (“Gangrène et nécrose des poumons”) n’était mappé qu’au concept 4324261 (“Nécrose pulmonaire”), qui n’est pas un descendant de “Trouble gangreneux.”
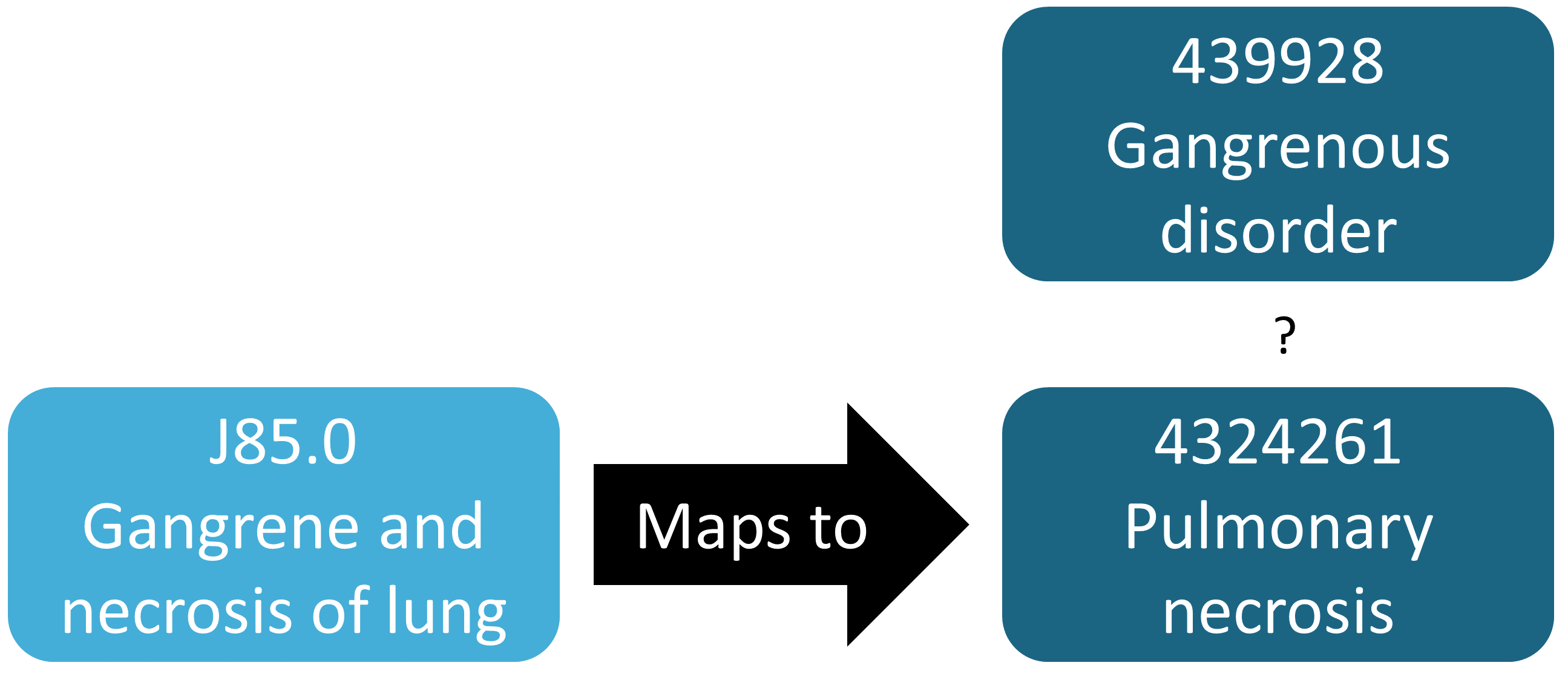
Figure 15.3: Example orphan source code.
15.3 ACHILLES en Pratique
Ici, nous allons démontrer comment exécuter ACHILLES contre une base de données au format CDM.
Nous devons d’abord dire à R comment se connecter au serveur. ACHILLES utilise le package DatabaseConnector, qui fournit une fonction appelée createConnectionDetails. Tapez ?createConnectionDetails pour les paramètres spécifiques requis pour les différents systèmes de gestion de bases de données (SGDB). Par exemple, on peut se connecter à une base de données PostgreSQL en utilisant ce code :
library(Achilles)
connDetails <- createConnectionDetails(dbms = "postgresql",
server = "localhost/ohdsi",
user = "joe",
password = "supersecret")
cdmDbSchema <- "my_cdm_data"
cdmVersion <- "5.3.0"Les deux dernières lignes définissent la variable cdmDbSchema, ainsi que la version CDM. Nous utiliserons ces informations plus tard pour dire à R où résident les données au format CDM, et quelle version de CDM est utilisée. Notez que pour Microsoft SQL Server, les schémas de base de données doivent spécifier à la fois la base de données et le schéma, donc par exemple cdmDbSchema <- "my_cdm_data.dbo".
Ensuite, nous exécutons ACHILLES:
result <- achilles(connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
resultsDatabaseSchema = cdmDbSchema,
sourceName = "My database",
cdmVersion = cdmVersion)Cette fonction créera plusieurs tables dans le resultsDatabaseSchema, que nous avons ici défini sur le même schéma de base de données que les données CDM.
Nous pouvons visualiser la caractérisation de la base de données ACHILLES. Cela peut être fait en pointant ATLAS vers les bases de données de résultats ACHILLES, ou en exportant les résultats ACHILLES vers un ensemble de fichiers JSON :
exportToJson(connectionDetails,
cdmDatabaseSchema = cdmDatabaseSchema,
resultsDatabaseSchema = cdmDatabaseSchema,
outputPath = "achillesOut")Les fichiers JSON seront écrits dans le sous-dossier achillesOut, et peuvent être utilisés avec l’application web AchillesWeb pour explorer les résultats. Par exemple, la Figure 15.4 montre le graphique de densité des données ACHILLES. Ce graphique montre que la majorité des données commence en 2005. Cependant, il semble aussi y avoir quelques enregistrements datant de 1961, ce qui est probablement une erreur dans les données.
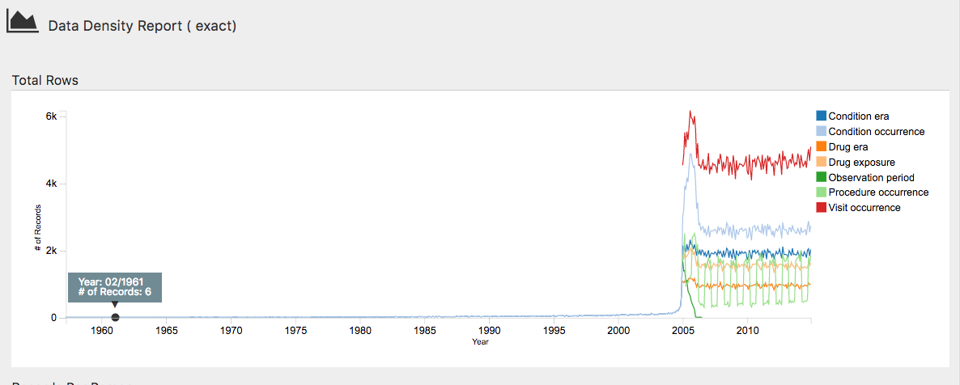
Figure 15.4: The data density plot in the ACHILLES web viewer.
Un autre exemple est montré dans la Figure 15.5, révélant un changement soudain dans la prévalence d’un code de diagnostic de diabète. Ce changement coïncide avec des modifications des règles de remboursement dans ce pays spécifique, entraînant plus de diagnostics mais probablement pas une véritable augmentation de la prévalence dans la population sous-jacente.
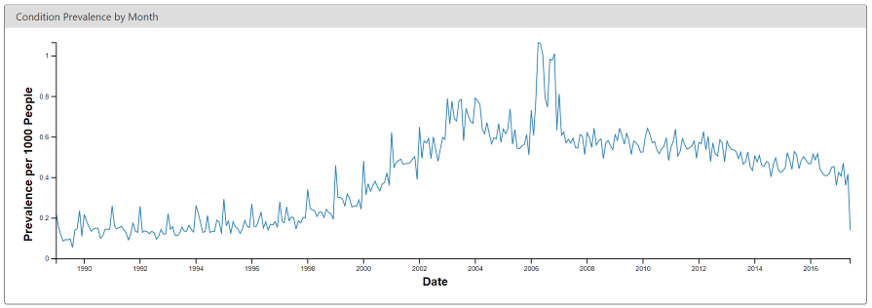
Figure 15.5: Monthly rate of diabetes coded in the ACHILLES web viewer.
15.4 Tableau de Bord de Qualité des Données en Pratique
Ici, nous allons démontrer comment exécuter le Tableau de Bord de Qualité des Données sur une base de données au format CDM. Nous le faisons en exécutant un grand ensemble de vérifications sur la connexion CDM décrite dans la Section 15.3. Pour l’instant, le DQD ne prend en charge que la version CDM v5.3.1, donc avant de vous connecter, assurez-vous que votre base de données est dans la bonne version. Comme avec ACHILLES, nous devons créer la variable cdmDbSchema pour indiquer à R où chercher les données.
Ensuite, nous exécutons le Tableau de Bord…
DataQualityDashboard::executeDqChecks(connectionDetails = connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
resultsDatabaseSchema = cdmDbSchema,
cdmSourceName = "My database",
outputFolder = "My output")La fonction ci-dessus exécutera toutes les vérifications de qualité des données disponibles sur le schéma spécifié. Elle écrira ensuite une table dans le resultsDatabaseSchema que nous avons ici défini comme le même schéma que le CDM. Cette table inclura toutes les informations sur chaque vérification effectuée, y compris la table CDM, le champ CDM, le nom de la vérification, la description de la vérification, la catégorie et la sous-catégorie Kahn, le nombre de lignes violées, le niveau de seuil, et si la vérification passe ou échoue, entre autres. En plus d’une table, cette fonction écrit également un fichier JSON à l’emplacement spécifié comme outputFolder. En utilisant ce fichier JSON, nous pouvons lancer un visualiseur web pour inspecter les résultats.
La variable jsonPath doit être le chemin vers le fichier JSON contenant les résultats du Tableau de Bord, situé dans le outputFolder spécifié lors de l’appel de la fonction executeDqChecks ci-dessus.
Lorsque vous ouvrez pour la première fois le Tableau de Bord, vous serez présenté(e) avec la table d’aperçu, comme illustré dans la Figure 15.6. Cela vous montrera le nombre total de vérifications effectuées dans chaque catégorie Kahn, réparties par contexte, le nombre et le pourcentage de succès dans chacune, ainsi que le taux global de réussite.
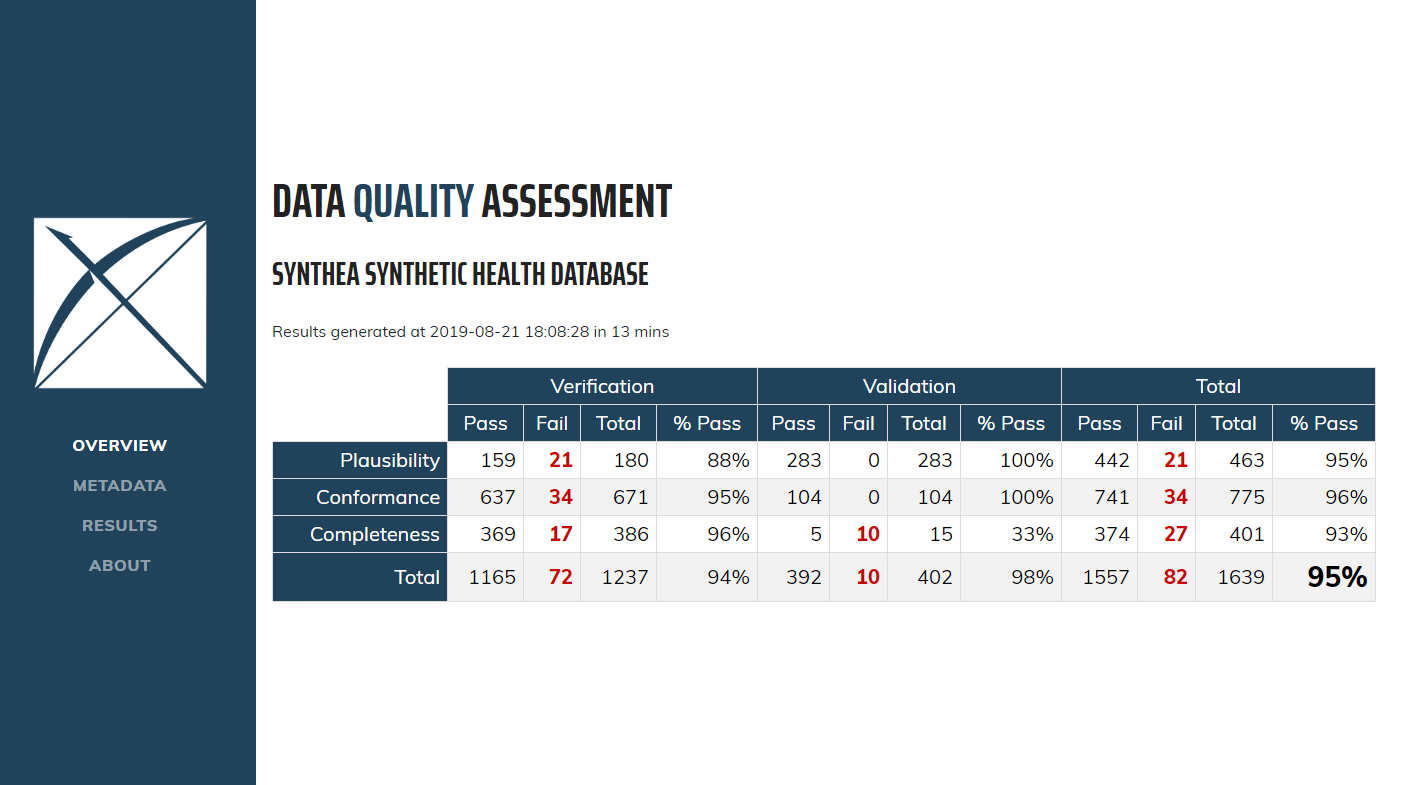
Figure 15.6: Overview of Data Quality Checks in the Data Quality Dashboard.
Cliquer sur Results dans le menu de gauche vous amènera aux résultats détaillés de chaque vérification effectuée (Figure 15.7). Dans cet exemple, la table montre une vérification effectuée pour déterminer la complétude des tables individuelles du CDM, ou, le nombre et le pourcentage de personnes dans le CDM avec au moins un enregistrement dans la table spécifiée. Dans ce cas, les cinq tables listées sont toutes vides, ce que le Tableau de Bord considère comme un échec. Cliquer sur l’icône ![]() ouvrira une fenêtre affichant la requête exacte qui a été exécutée sur vos données pour produire les résultats listés. Cela permet d’identifier facilement les lignes considérées comme des échecs par le Tableau de Bord.
ouvrira une fenêtre affichant la requête exacte qui a été exécutée sur vos données pour produire les résultats listés. Cela permet d’identifier facilement les lignes considérées comme des échecs par le Tableau de Bord.
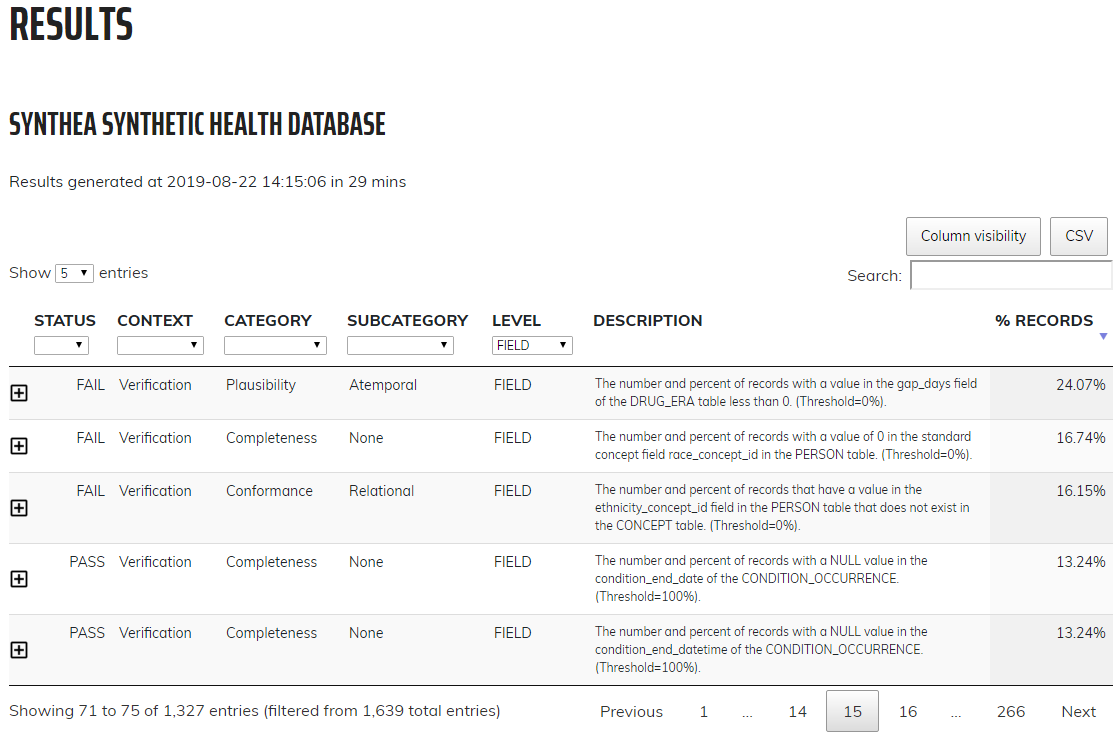
Figure 15.7: Drilldown into Data Quality Checks in the Data Quality Dashboard.
15.5 Vérifications Spécifiques à l’Étude en Pratique
Ensuite, nous exécuterons plusieurs vérifications spécifiquement pour la définition de cohorte angioedème fournie dans l’Appendice 22.4. Nous supposerons que les détails de connexion ont été définis comme décrit dans la Section 15.3, et que le JSON et le SQL de la définition de la cohorte ont été enregistrés dans les fichiers “cohort.json” et “cohort.sql”, respectivement. Les fichiers JSON et SQL peuvent être obtenus à partir de l’onglet Exporter dans la fonction de définition de cohorte d’ATLAS.
library(MethodEvaluation)
json <- readChar("cohort.json", file.info("cohort.json")$size)
sql <- readChar("cohort.sql", file.info("cohort.sql")$size)
checkCohortSourceCodes(connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
cohortJson = json,
cohortSql = sql,
outputFile = "output.html")Nous pouvons ouvrir le fichier de sortie dans un navigateur web comme illustré dans la Figure 15.8. Ici, nous voyons que la définition de la cohorte angioedème a deux ensembles de concepts : “Visite en hospitalisation ou aux urgences” et “Angioedème”. Dans cet exemple de base de données, les visites ont été trouvées par des codes sources spécifiques à la base de données “ER” et “IP”, qui ne sont pas dans le Vocabulaire, bien qu’elles aient été mappées lors de l’ETL à des concepts standard. Nous voyons également que l’angioedème est détecté par un code ICD-9 et deux codes ICD-10. Nous voyons clairement le point dans le temps de la transition entre les deux systèmes de codage lorsque nous regardons les mini-graphes pour les codes individuels, mais pour l’ensemble de concepts dans son ensemble, il n’y a pas de discontinuité à ce moment-là.
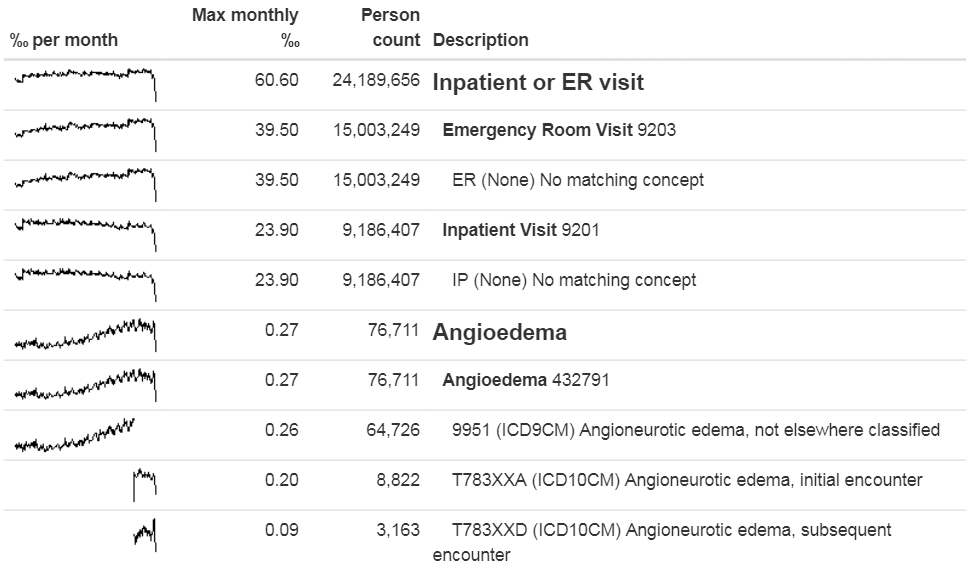
Figure 15.8: Source codes used in the angioedema cohort definition.
Ensuite, nous pouvons rechercher des codes sources orphelins, ce qui sont des codes sources qui ne sont pas mappés à des codes de concepts standard. Ici, nous recherchons le Concept Standard “Angioedème”, puis nous recherchons tous les codes et concepts qui ont “Angioedème” ou l’un des synonymes que nous fournissons dans leur nom :
orphans <- findOrphanSourceCodes(connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
conceptName = "Angioedema",
conceptSynonyms = c("Angioneurotic edema",
"Giant hives",
"Giant urticaria",
"Periodic edema"))
View(orphans)| code | description | vocabularyId | overallCount |
|---|---|---|---|
| T78.3XXS | Angioneurotic edema, sequela | ICD10CM | 508 |
| 10002425 | Angioedemas | MedDRA | 0 |
| 148774 | Angioneurotic Edema of Larynx | CIEL | 0 |
| 402383003 | Idiopathic urticaria and/or angioedema | SNOMED | 0 |
| 232437009 | Angioneurotic edema of larynx | SNOMED | 0 |
| 10002472 | Angioneurotic edema, not elsewhere classified | MedDRA | 0 |
Le seul orphelin potentiel trouvé qui est effectivement utilisé dans les données est “Angioneurotic edema, sequela”, qui ne devrait pas être mappé à angioedème. Cette analyse n’a donc révélé aucun code manquant.
15.6 Résumé
La plupart des données de soins de santé observationnelles n’ont pas été collectées à des fins de recherche.
Les contrôles de qualité des données sont une partie intégrante de la recherche. La qualité des données doit être évaluée pour déterminer si les données sont de qualité suffisante pour les besoins de la recherche.
Nous devons évaluer la qualité des données à des fins de recherche en général, et de manière critique dans le contexte d’une étude spécifique.
Certains aspects de la qualité des données peuvent être évalués automatiquement grâce à de grands ensembles de règles prédéfinies, par exemple celles du Tableau de Bord de Qualité des Données.
D’autres outils existent pour évaluer la cartographie des codes pertinents pour une étude particulière.
15.7 Exercices
Prérequis
Pour ces exercices, nous supposons que R, R-Studio et Java ont été installés comme décrit dans la Section 8.4.5. Sont également nécessaires les packages SqlRender, DatabaseConnector, ACHILLES, et Eunomia, qui peuvent être installés en utilisant :
install.packages(c("SqlRender", "DatabaseConnector", "remotes"))
remotes::install_github("ohdsi/Achilles")
remotes::install_github("ohdsi/DataQualityDashboard")
remotes::install_github("ohdsi/Eunomia", ref = "v1.0.0")Le package Eunomia fournit un ensemble de données simulé dans le CDM qui s’exécutera dans votre session R locale. Les détails de la connexion peuvent être obtenus à l’aide de :
Le schéma de base de données CDM est “main”.
Exercice 15.1 Exécutez ACHILLES sur la base de données Eunomia.
Exercice 15.2 Exécutez le Tableau de Bord de Qualité des Données sur la base de données Eunomia.
Exercice 15.3 Extrayez la liste des vérifications du DQD.
Les réponses suggérées se trouvent dans l’appendice 24.9.