第 15 章 データ品質
著者: Martijn Schuemie, Vojtech Huser & Clair Blacketer
医療観察研究に用いられるデータのほとんどは、研究目的で収集されたものではない。例えば、電子的健康記録(EHR)は患者のケアをサポートするために必要な情報を捕捉することを目的としており、保険請求データは保険者への費用配分を割り当てるために収集されています。このようなデータを臨床研究に用いることが適切であるかどうかについては、多くの疑問が呈されています。Lei (1991) は「データは収集された目的のみに使用されるべきである」とさえ述べています。懸念されるのは、私たちがやりたい研究のために収集されたデータではないため、そのデータの質が十分である保証がないということです。データの品質が低い(入力がゴミ)場合、そのデータを使用した研究結果の質も低い(出力もゴミ)に違いないでしょう。したがって、観察医療研究の重要な側面はデータの質を評価することであり、次の質問に答えることを目指しています。
研究目的に対して、データの品質は十分でしょうか?
データ品質(DQ)を次のように定義できます(Roebuck 2012):
特定の使用目的に適したデータとなるような、完全性、妥当性、一貫性、適時性、正確性。
データが完璧であることはまずありませんが、目的には十分である可能性があることに留意ください。
データ品質は直接観察することができませんが、それを評価する方法論が開発されています。データ品質評価には2つの種類があります(Weiskopf and Weng 2013):データ品質を全般的に評価する評価と、特定の研究におけるデータ品質を評価する評価です。
本章では、まずデータ品質の問題の原因となり得るものを検討し、その後、一般的なデータ品質評価と特定の研究におけるデータ品質評価の理論について説明し、最後に、OHDSIツールを使用してこれらの評価を実行する方法を段階的に説明します。
15.1 データ品質問題の原因
第14章で述べたように、医師が自身の考えを記録する段階からデータの品質に対する脅威は数多く存在します。Dasu and Johnson (2003) は、データのライフサイクルにおいて次のステップを区別し、各ステップにデータ品質を統合することを推奨しています。これをデータ品質の連続体と呼んでいます。
- データ収集と統合。考えられる問題としては、手入力の誤り、バイアス(例: 保険請求におけるコーディングの誤り)、EHRでのテーブルの誤った結合、欠測値のデフォルト値への置き換えなどがあります。
- データの保存と知識の共有。考えられる問題としては、データモデルの文書化不足やメタデータの欠如などが挙げられます。
- データ分析。不正確なデータ変換、データの誤った解釈、不適切な方法論の使用などなどの問題が含まれます。
- データの公開。下流での使用のためにデータを公開する際の問題。
私たちが使用するデータはすでに収集され統合されていることが多いため、ステップ1を改善するためにできることはほとんどありません。この章の後半で詳しく述べるように、このステップによって生成されるデータ品質をチェックする方法があります。
同様に、私たちは特定の形式でデータを頻繁に受け取っているため、ステップ2の一部についてはほとんど影響力を持ちません。しかし、OHDSIでは、すべての観測データを共通データモデル(CDM)に変換しており、このプロセスについては私たちが管理権限を持っています。この特定のステップがデータ品質を低下させる可能性があるという懸念が示されたこともあります。しかし、私たちがこのプロセスを管理しているため、後述のセクション 15.2.2 で説明するように、データ品質を維持するための厳格な保護策を構築することができます。いくつかの調査(Defalco, Ryan, and Soledad Cepeda 2013; Makadia and Ryan 2014; Matcho et al. 2014; Voss, Ma, and Ryan 2015; Voss et al. 2015; G. Hripcsak et al. 2018)によれば、正しく実行されれば、CDMへの変換時にほとんどエラーは発生しないことが示されています。実際、大規模なコミュニティで共有される詳細に文書化されたデータモデルがあれば、曖昧さのない明確な方法でデータの保存が容易になります。
ステップ3(データ分析)もまた、私たちの管理下にあります。OHDSIでは、このステップにおける品質問題についてデータ品質という用語は使用せず、臨床妥当性、ソフトウェア妥当性、方法妥当性という用語を使用しています。これらの用語については、それぞれ第16、17、18章で詳しく議論しています。
15.2 一般的なデータ品質
観察研究の一般的な目的に対してデータが適しているかどうかを問うことができます。Michael G. Kahn et al. (2016) は、このような一般的なデータ品質を次の3つの要素から構成されるものと定義しています。
適合性:データ値が指定された基準や形式に従っているでしょうか。3つのサブタイプに識別されます:
- 値:記録されたデータ要素が指定された形式に合致しているでしょうか。例えば、すべての医療専門分野は有効な専門分野でしょうか。
- 関係:記録されたデータが指定された関係制約に合致しているでしょうか。例えば、DRUG_EXPOSURE データの PROVIDER_ID が PROVIDER テーブルの対応するレコードと一致しているでしょうか。
- 計算:データに対する計算結果が意図したとおりになっているでしょうか。例えば、身長と体重から計算されたBMIがデータに記録されているものと等しいでしょうか。
完全性:特定の変数が存在するかどうか(例:診察室で測定された体重が記録されているか?)や、変数がすべての記録された値を含んでいるか(例:すべての人の性別が分かっているか)を参照します。
妥当性(plausibility):データ値は信頼できるでしょうか。3つのサブタイプが定義されています:
- 一意性:例えば、PERSON テーブルで各 PERSON_ID は一度しか出現しないでしょうか?
- 非時間的:値、分布、密度が期待される値と一致しているでしょうか?例えば、データから示唆される糖尿病の有病率は既知の有病率と一致しているでしょうか?
- 時間的:値の変化は期待と一致しているでしょうか?例えば、予防接種の順序は推奨事項と一致しているでしょうか。
各コンポーネントは2つの方法で評価できます:
- 検証 では、モデルとメタデータのデータ制約、システムの前提条件、ローカルの知識に焦点を当てます。外部参照には依存しません。検証の主な特徴は、ローカル環境内のリソースを使用して、期待される値と分布を決定する能力です。
- 妥当性(バリデーション) では、関連する外部ベンチマークとのデータ値の整合性に焦点を当てます。外部ベンチマークのソースの1つとして、複数のデータサイトにわたる結果を組み合わせることが考えられます。
15.2.1 データ品質チェック
カーンは、データが所定の要件に適合しているかどうかをテストするデータ品質チェック(データ品質ルールと呼ばれることもある)という用語を導入しています(例えば、患者の年齢が141歳というありえない値になっている場合、誤った生年が入力されたか、死亡イベントが記録されていない可能性があります)。このようなチェックは、自動化されたデータ品質ツールを作成することでソフトウェアに実装することができます。そのようなツールの1つに、ACHILLES(Automated Characterization of Health Information at Large-scale Longitudinal Evidence Systems)があります(Huser et al. 2018)。ACHILLESは、CDMに準拠したデータベースの特性評価と可視化を行うソフトウェアツールです。そのため、データベースのネットワークにおけるDQの評価に使用することができます(Huser et al. 2016)。ACHILLESはスタンドアロンツールとして利用でき、「データソース」機能としてもATLASに統合されています。
ACHILLESは、170以上のデータ特性評価を事前に計算します。各分析には分析IDと分析の簡単な説明があり、その例として「715:DRUG_CONCEPT_IDによるDAYS_SUPPLYの分布」や「506:性別による死亡時の年齢の分布」などがあります。これらの分析結果はデータベースに保存され、ウェブビューアーまたはATLASからアクセスできます。
コミュニティが作成したもう一つのツールで、DQ を評価するものに、Data Quality Dashboard (DQD) があります。ACHILLES が特性評価を実行して CDM インスタンスの全体像を視覚的に把握できるようにするのに対し、DQD は表ごとに、またフィールドごとに、CDM 内の所定の仕様を満たさないレコード数を数値化します。合計で 1,500 を超えるチェックが実行され、それぞれが Kahn のフレームワークで整理されています。各チェックの結果は閾値と比較され、違反行の割合がその値を上回る場合は不合格とみなされます。表 15.1 は、いくつかのチェックの例を示しています。
| 違反行の割合 | チェックの説明 | 閾値 | 状態 |
|---|---|---|---|
| 0.34 | VISIT_OCCURRENCE の provider_id が仕様に基づく期待されるデータ型であるかどうかを示す yes、 no の値。 | 0.05 | FAIL |
| 0.99 | MEASUREMENT テーブルの measurement_source_value フィールドにある異なるソース値の数やパーセントが 0 にマッピングされている。 | 0.30 | FAIL |
| 0.09 | DRUG_ERA テーブルの drug_concept_id フィールドにある値が成分クラスに適合しない記録の数、パーセント。 | 0.10 | PASS |
| 0.02 | DRUG_EXPOSURE テーブルの DRUG_EXPOSURE_END_DATE フィールドの値が DRUG_EXPOSURE_START_DATE フィールドの日付より前に発生する記録の数、パーセント。 | 0.05 | PASS |
| 0.00 | PROCEDURE_OCCURRENCE テーブルの procedure_occurrence_id フィールドに重複する値がある記録の数、パーセント。 | 0.00 | PASS |
このツールでは、チェックは複数の方法で整理されており、その一つはテーブル、フィールド、コンセプトレベルのチェックです。テーブルレベルのチェックは、CDM内の高次レベルで行われるもので、例えば、必要なテーブルがすべて揃っているかどうかの判断などです。フィールドレベルのチェックは、CDMの仕様への適合性を評価するために、各テーブル内のすべてのフィールドに対して実施されます。これには、すべての主キーが実際に一意であること、すべての標準コンセプトフィールドが適切なドメインにコンセプトIDを含んでいることなど、多くの項目が含まれます。コンセプトレベルのチェックは、個々のコンセプトIDをさらに詳しく検証して実施ます。これらの多くは、Kahnフレームワークの妥当性カテゴリーに分類されます。例えば、性別特有のコンセプトが誤った性別の人物に帰属されていないこと(すなわち、女性患者の前立腺癌)を確認することなどです。
15.2.2 ETL(抽出-変換-読込)単体テスト
高度なデータ品質のチェックに加え、個別レベルでのデータチェックも実施すべきです。データがCDMに変換されるETLプロセスは、非常に複雑であることがよくあり、その複雑さゆえに、気づかれないままミスが発生する危険性があります。さらに、時間の経過とともにソースデータモデルが変更されたり、CDMが更新されたりすることもあり、ETLプロセスを修正する必要が生じます。ETL のように複雑なプロセスに変更を加えると予期せぬ結果を招く可能性があり、ETL のすべての側面を再考し、再評価する必要が生じます。
ETL が期待通りに動作し、その状態を維持できるようにするためには、一連の単体テストを作成することが強く推奨されます。単体テストとは、自動的に単一の側面をチェックする小さなコードの断片です。第6章で説明したRabbit-in-a-Hatツールを使用すると、このような単体テストを簡単に作成できるユニットテストフレームワークを作成することができます。このフレームワークは、ソースデータベースとターゲットCDMバージョンのETL用に特別に作成されたR関数の集合です。これらの関数の一部は、ソースデータスキーマに準拠した偽のデータエントリを作成するためのもので、その他の関数はCDM形式のデータに関する期待値を指定するために使用できます。以下に単体テストの例を示します。
source("Framework.R")
declareTest(101, "Person gender mappings")
add_enrollment(member_id = "M000000102", gender_of_member = "male")
add_enrollment(member_id = "M000000103", gender_of_member = "female")
expect_person(PERSON_ID = 102, GENDER_CONCEPT_ID = 8507
expect_person(PERSON_ID = 103, GENDER_CONCEPT_ID = 8532)この例では、Rabbit-in-a-Hatによって生成されたフレームワークがソースとして読み込まれ、コードの残りの部分で使用される関数が読み込まれます。次に、人物の性別マッピングのテストを開始することを宣言します。ソース・スキーマにはENROLLMENTテーブルがあり、Rabbit-in-a-Hatによって作成されたadd_enrollment関数を使用して、MEMBER_IDおよびGENDER_OF_MEMBERフィールドに異なる値を持つ2つのエントリを作成します。最後に、ETL 実行後には、さまざまな期待値を持つ 2 つのエントリが PERSON テーブルに存在しているはずであるという期待値を指定します。
ENROLLMENT テーブルには他にも多くのフィールドがありますが、このテストのコンテキストでは、それらのフィールドの値についてはあまり気にする必要はありません。ただし、それらの値(生年月日など)を空のままにしておくと、ETL がレコードを破棄したりエラーを発生させたりする可能性があります。この問題を克服し、テストコードを読みやすく保つために、add_enrollment関数は、ユーザーによって明示的に指定されていないフィールド値にデフォルト値(White Rabbit スキャンレポートで観測された最も一般的な値)を割り当てます。
同様のユニットテストをETLの他のすべてのロジックに対して作成することもでき、通常は数百のテストが作成されます。テストの定義が完了したら、フレームワークを使用して2つのSQLステートメントセットを生成します。1つは偽のソースデータを作成し、もう1つはETL化されたデータに対するテストを作成します。
insertSql <- generateInsertSql(databaseSchema = "source_schema")
testSql <- generateTestSql(databaseSchema = "cdm_test_schema")全体のプロセスは図 15.1 に示されています。
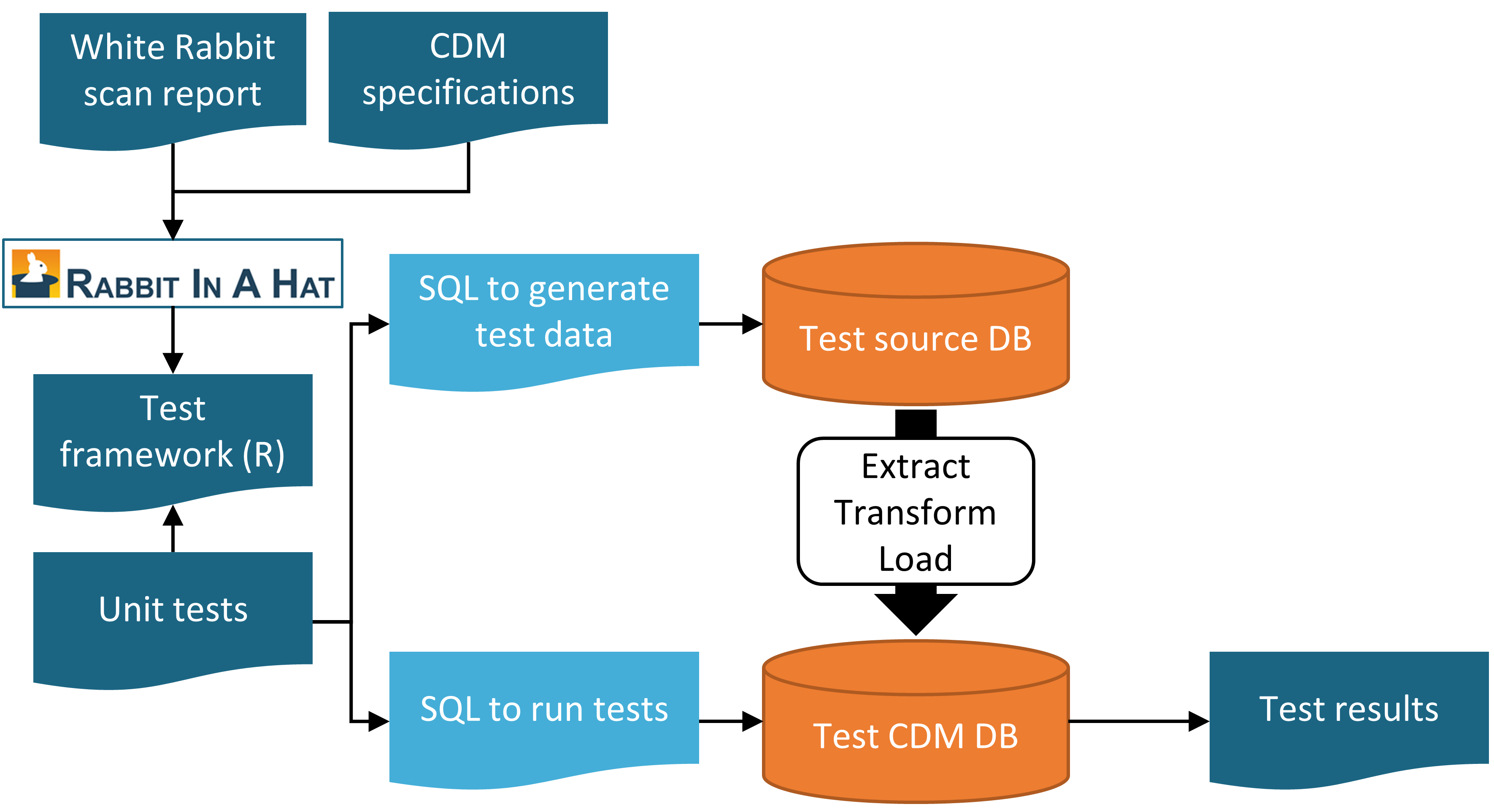
図 15.1: Rabbit-in-a-Hatテストフレームワークを使用したETL(抽出-変換-読込)プロセスの単体テスト
テスト用のSQLは、表 15.2 のようなテーブルを返します。このテーブルでは、先に定義した2つのテストに合格したことがわかります。
| ID | 説明 | 状態 |
|---|---|---|
| 101 | Person gender mappings | PASS |
| 101 | Person gender mappings | PASS |
これらの単体テストの力は、ETL プロセスが変更されたときに簡単に再実行できることです。
15.3 研究特有のチェック
この章では、これまでは一般的なDQチェックに焦点を当ててきました。このようなチェックは、データを研究に用いる前に実行すべきです。これらのチェックは調査の質問とは関係なく実行されるため、研究固有のDQ評価を行うことを推奨しています。
これらの評価の一部は、調査に特に関連するDQルールという形式を取ることができます。例えば、関心のある曝露に関するレコードの少なくとも90%が曝露期間を指定しているというルールを課すことが考えられます。
標準的な評価は、ACHILLESで研究に最も関連するコンセプトを検討することであり、例えば、コホート研究の定義で指定されたものなどがあります。コードが観察される割合が時間とともに突然変化する場合は、DQの問題が示唆される場合があります。いくつかの例は、この章で後ほど説明します。
別の評価方法としては、研究用に開発されたコホート定義を使用して生成されたコホートの有病率や経時的な有病率の変化を再検討し、それらが外部臨床知識に基づく予測と一致しているかどうかを確認することが挙げられます。例えば、新薬への曝露は市場に投入される前には存在しないはずであり、投入後は時間の経過とともに増加する可能性が高いです。同様に、アウトカムの有病率は、その集団における疾患の有病率として知られている内容と一致しているはずです。研究がデータベースのネットワーク全体で実施された場合、データベース間でコホートの有病率を比較することができます。あるデータベースではコホートの有病率が非常に高いが、別のデータベースでは欠落している場合、DQの問題がある可能性があります。このような評価は、第16章で説明されているように、臨床的妥当性のコンセプトと重複していることに留意ください。一部のデータベースで予期せぬ有病率が見られるのは、DQの問題ではなく、コホートの定義が、対象とする疾患状態を正確に捉えていないため、あるいは、異なる患者集団を捉えているデータベース間で、これらの健康状態が当然異なるためである可能性があります。
15.3.1 マッピングのチェック
私たちの管理下で明確に該当するエラーの可能性として、ソースコードから標準コンセプトへのマッピングが挙げられます。 ボキャブラリのマッピングは入念に作成されており、コミュニティのメンバーによって指摘されたマッピングのエラーは、ボキャブラリのイシュー追跡システム57に報告され、今後のリリースで修正されます。しかし、すべてのマッピングを手作業で完全にチェックすることは不可能であり、エラーが残っている可能性は依然としてあります。そのため、調査を行う際には、その調査に最も関連性の高いコンセプトのマッピングを確認することをお勧めします。幸い、これは非常に簡単に実行できます。なぜなら、CDMでは標準コンセプトだけでなくソースコードも保存されているからです。研究で使用されたコンセプトにマッピングされたソースコードだけでなく、そうでないソースコードも確認することができます。
対応づけられたソースコードをレビューする方法のひとつに、RパッケージMethodEvaluationのcheckCohortSourceCodes関数を使用する方法があります。この関数は、ATLASによって作成されたコホート定義を入力として使用し、コホート定義で使用されている各コンセプトセットについて、そのセット内のコンセプトに対応づけられたソースコードがどれであるかをチェックします。また、特定のソースコードに関連する時間的な問題を特定するのに役立つよう、これらのコードの経時的な有病率も計算します。図 15.2 の出力例は、「うつ病性障害」と呼ばれるコンセプトセットの一部内訳を示しています。 対象のデータベースにおけるこのコンセプトセットで最も頻度の高いコンセプトは、コンセプト440383(「うつ病性障害」)です。 このデータベースでは、ICD-9コード3.11、ICD-10コードF32.8、F32.89の3つのソースコードがこのコンセプトにマッピングされていることが分かります。左側では、このコンセプト全体がまず徐々に増加し、その後急激に減少していることが分かります。個々のコードを調べると、この減少は、減少の時期にICD-9コードが使用されなくなったことによるものであることが分かります。これはICD-10コードが使用され始めた時期と一致していますが、ICD-10コードの合計の有病率はICD-9コードの有病率よりもはるかに低くなっています。この特定の例は、ICD-10コードのF32.9(「うつ病、単一エピソード,詳細不明」)もまた、このコンセプトにマッピングされるべきであったという事実によるものです。この問題は、その後、ボキャブラリで解決されました。
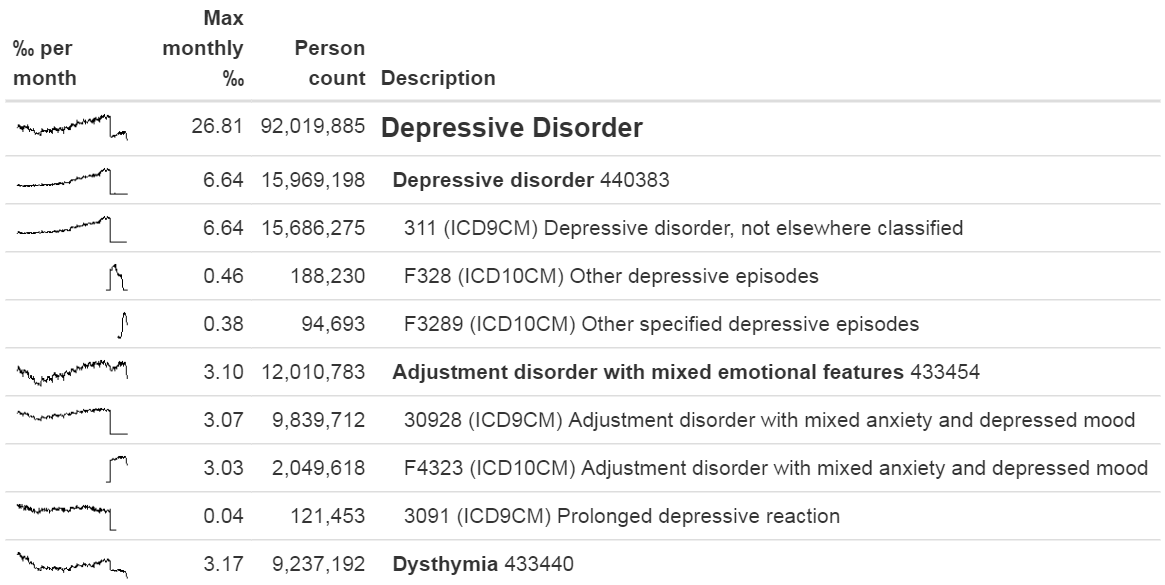
図 15.2: checkCohortSourceCodes関数のサンプル出力
前述の例では、マッピングされていないソースコードが偶然発見されたことを示していますが、一般的に、存在するマッピングの確認よりも、欠落しているマッピングの特定の方が困難です。どのソースコードがマッピングされるべきだが、されていないのかを知る必要があります。この評価を半自動的に行う方法としては、MethodEvaluation RパッケージのfindOrphanSourceCodes関数を使用する方法があります。この関数を使用すると、簡単なテキスト検索でボキャブラリの中からソースコードを検索し、それらのソースコードが特定のコンセプトまたはそのコンセプトの下位層のいずれかにマッピングされているかどうかを確認することができます。 結果として得られたソースコードのセットは、次に、手元のCDMデータベースに表示されているものだけに制限されます。 例えば、研究では「壊疽性疾患」(439928)というコンセプトと、その下位層すべてを使用して、壊疽のすべての出現箇所を特定しました。この検索結果が、実際に壊疽を示すすべてのソースコードを含んでいるかどうかを評価するために、いくつかの用語(例えば「壊疽」)を使用して、ソースコードを特定するためにコンセプトとSOURCE_TO_CONCEPT_MAPテーブルの説明を検索しました。次に、自動検索を使用して、データに表示される各壊疽ソースコードが、実際に直接または間接的(上位層経由)にコンセプト「壊疽性疾患」にマッピングされているかどうかを評価しました。この評価の結果は図 15.3 に示されています。ICD-10のJ85.0(「肺壊疽および壊死」)は、4324261(「肺壊死」)というコンセプトにのみマッピングされており、これは「壊疽性障害」の下位層ではありません。
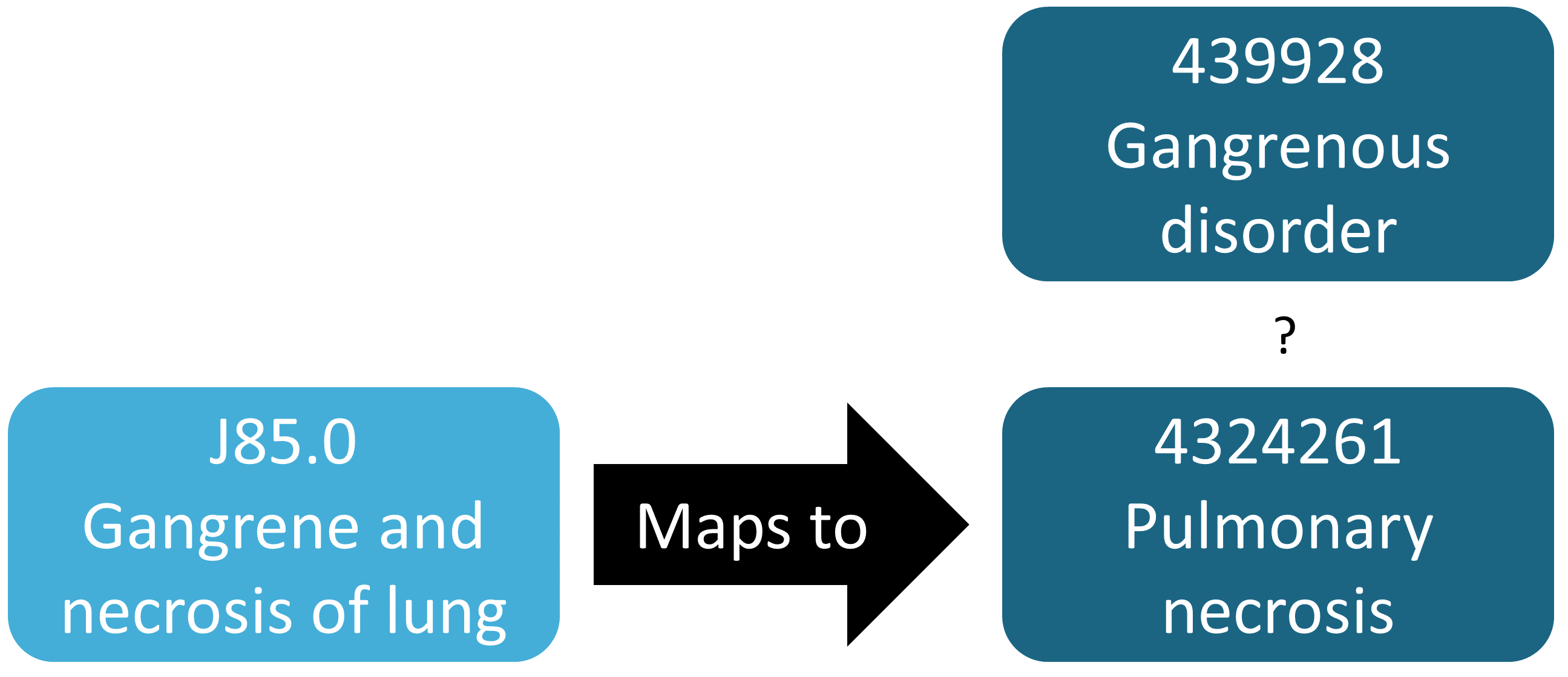
図 15.3: 孤立したソースコードの例
15.4 実践におけるACHILLES
ここでは、CDM形式のデータベースに対してACHILLESを実行する方法を説明します。
まず、Rにサーバーへの接続方法を指示する必要があります。ACHILLESは、DatabaseConnectorパッケージを使用しており、このパッケージはcreateConnectionDetailsと呼ばれる関数を提供しています。createConnectionDetailsと入力すると、さまざまなデータベース管理システム(DBMS)に必要な特定の設定を行うことができます。例えば、以下のコードを使用してPostgreSQLデータベースに接続することができます。
library(Achilles)
connDetails <- createConnectionDetails(dbms = "postgresql",
server = "localhost/ohdsi",
user = "joe",
password = "supersecret")
cdmDbSchema <- "my_cdm_data"
cdmVersion <- "5.3.0"最後の2行では、cdmDbSchema変数とCDMのバージョンを定義しています。 これらは、CDM形式のデータがどこに存在し、どのバージョンのCDMが使用されているかをRに伝えるために使用します。Microsoft SQL Serverでは、データベーススキーマではデータベースとスキーマの両方を指定する必要があることに留意ください。例えば、cdmDbSchema <- 「my_cdm_data.dbo」となります。
次に、ACHILLESを実行します:
result <- achilles(connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
resultsDatabaseSchema = cdmDbSchema,
sourceName = "My database",
cdmVersion = cdmVersion)この関数は、resultsDatabaseSchema内に複数のテーブルを作成します。ここでは、CDMデータと同じデータベーススキーマに設定しています。 ACHILLESデータベース特性評価を表示することができます。これは、ATLASをACHILLES結果データベースにポイントするか、ACHILLES結果をJSONファイルのセットにエクスポートすることで実行できます。
exportToJson(connectionDetails,
cdmDatabaseSchema = cdmDatabaseSchema,
resultsDatabaseSchema = cdmDatabaseSchema,
outputPath = "achillesOut")JSONファイルはachillesOutサブフォルダに書き込まれ、AchillesWebウェブアプリケーションと併用して結果を調査することができます。例えば、図 15.4 はACHILLESデータ密度プロットを示しています。このプロットは、データの大部分が2005年から始まっていることを示しています。しかし、1961年頃のレコードもいくつか存在しているように見えますが、これはおそらくデータのエラーです。
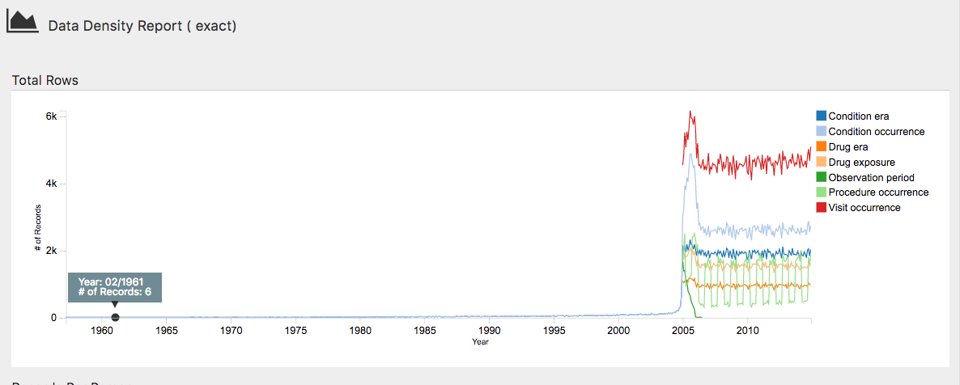
図 15.4: ACHILLESウエブビューワーでのデータ密度プロット
別の例を図 15.5 に示します。これは、糖尿病の診断コードの有病率に急激な変化が生じていることを示しています。この変化は、この特定の国における償還規則の変更と時期が一致しており、より多くの診断につながっていますが、おそらく基礎となる集団における真の有病率の増加ではないでしょう。
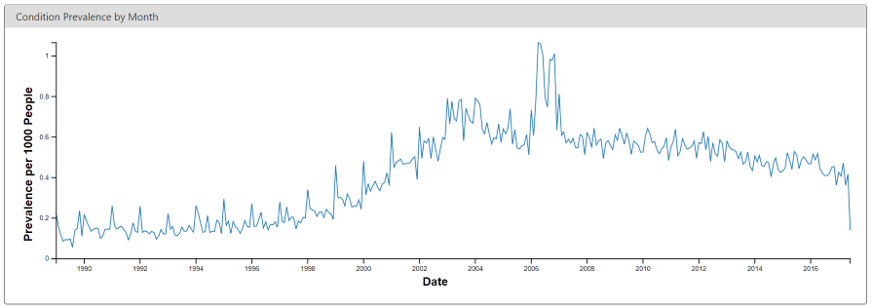
図 15.5: ACHILLESウエブビューワーでの月次糖尿病の診断コードの有病率
15.5 Data Quality Dashboardの実践
ここでは、CDM 形式のデータベースに対してデータ品質ダッシュボード(Data Quality Dashboard;DQD)を実行する方法を説明します。 これを行うには、セクション 15.4 で説明されているCDM接続に対して、一連のチェックを実行します。 現時点では、DQDはCDM v5.3.1のみをサポートしているため、接続する前にデータベースが正しいバージョンであることを確認してください。 ACHILLESの場合と同様に、Rにデータの検索先を指示するために、変数 cdmDbSchema を作成する必要があります。
次に、DQDを実行します…
DataQualityDashboard::executeDqChecks(connectionDetails = connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
resultsDatabaseSchema = cdmDbSchema,
cdmSourceName = "My database",
outputFolder = "My output")上記の関数は、指定されたスキーマ上で利用可能なすべてのデータ品質チェックを実行します。その後、resultsDatabaseSchemaにテーブルを書き込みます。ここでは、CDMと同じスキーマに設定しています。このテーブルには、CDMテーブル、CDMフィールド、チェック名、チェックの説明、Kahnのカテゴリーおよびサブカテゴリー、違反行の数、閾値レベル、チェックの合否など、各チェック実行に関するすべての情報が含まれます。この関数は、テーブルに加えて、outputFolderとして指定された場所にJSONファイルも書き込みます。このJSONファイルを使用して、結果を検査するためのウェブビューアーを起動することができます。
変数jsonPathは、上記のexecuteDqChecks関数を呼び出す際に指定したoutputFolderにある、ダッシュボードの結果を含むJSONファイルへのパスである必要があります。
最初にダッシュボードを開くと、図 15.6 に示すような概要テーブルが表示されます。このテーブルには、コンテキスト別に分類された各Kahnカテゴリで実行されたチェックの合計数、それぞれの合格数と合格率、および全体の合格率が表示されます。
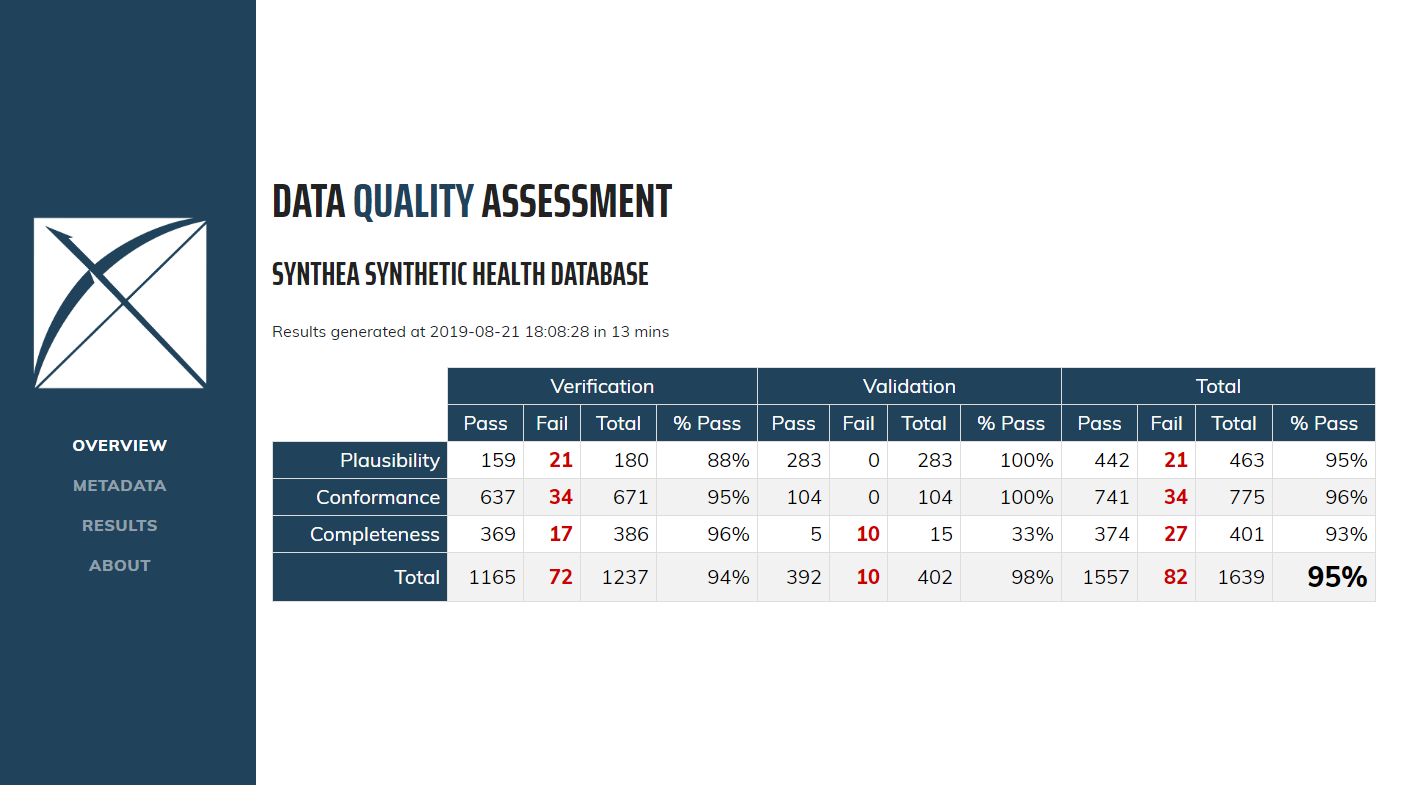
図 15.6: Data Quality Dashboardにおけるデータ品質チェックの概要
左側のメニューでResults(結果)をクリックすると、実行された各チェックのドリルダウン結果が表示されます(図 15.7)。この例では、個々のCDMテーブルの完全性、すなわち、指定されたテーブルに少なくとも1つのレコードを持つCDM内の人数とパーセンテージを決定するために実行されたチェックを示す表が表示されます。この場合、ダッシュボードは5つのテーブルがすべて空であるとカウントし、失敗と見なします。アイコンをクリックすると、リストされた結果を生成するためにデータに対して実行された正確なクエリを表示するウィンドウが開きます。これにより、ダッシュボードでエラーとみなされた行を簡単に特定できます。
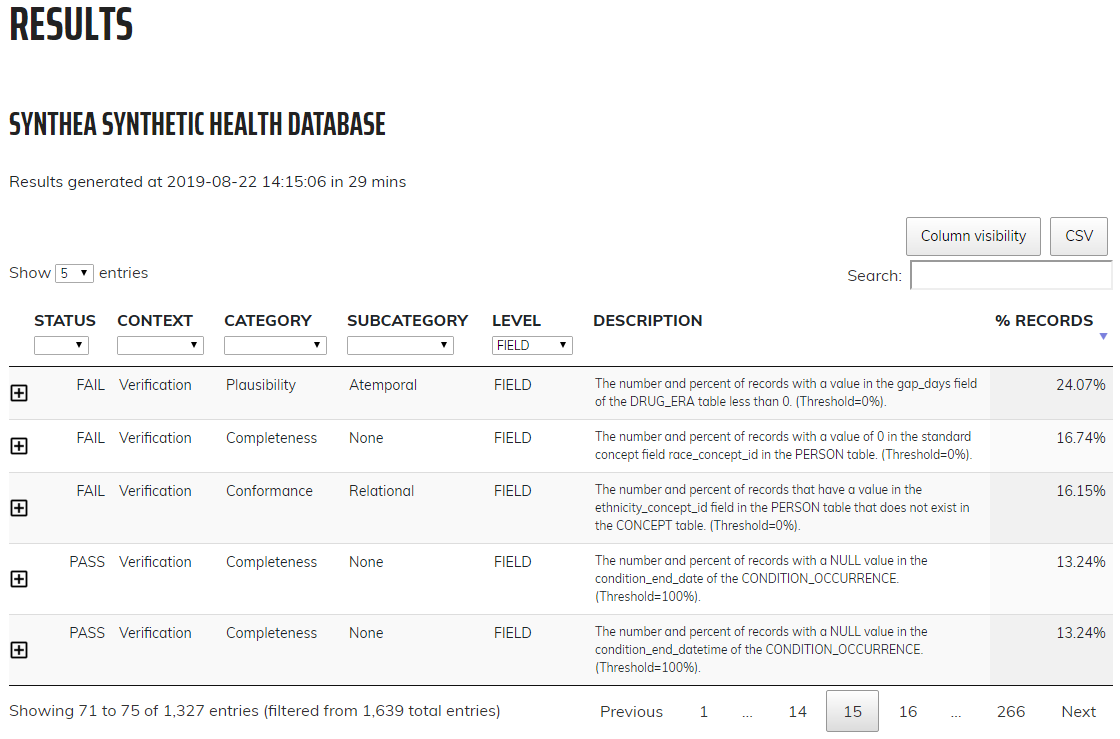
図 15.7: Data Quality Dashboardにおけるデータ品質チェックの詳細
15.6 特定の研究チェックの実践
次に、付録 B.4 で提供されている血管性浮腫コホート定義に特化したいくつかのチェックを実行します。接続の詳細はセクション 15.4 で説明されているように設定済みであり、コホート定義のJSONとSQLはそれぞれ「cohort.json」と「cohort.sql」というファイルに保存済みであると仮定します。JSONとSQLは、ATLASコホート定義機能のエクスポートタブから取得できます。
library(MethodEvaluation)
json <- readChar("cohort.json", file.info("cohort.json")$size)
sql <- readChar("cohort.sql", file.info("cohort.sql")$size)
checkCohortSourceCodes(connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
cohortJson = json,
cohortSql = sql,
outputFile = "output.html")出力ファイルをウェブブラウザで開くことができます(図 15.8)。ここでは、血管性浮腫のコホート定義には「Inpatient or ER(入院または救急室ビジット)」と「Angioedema(血管性浮腫)」という二つのコンセプトセットがあります。この例のデータベースでは、ビジットは「ER」および「IP」というデータベース固有のソースコードによって同定され、ETL中に標準コンセプトにマッピングされました。また、血管性浮腫は一つのICD-9コードと二つのICD-10コードによって同定されました。それぞれのコードのスパークラインを見ると、二つのコーディングシステムのどの時点で切り替わったかが明らかになりますが、コンセプトセット全体としてはその時点での不連続性はありません。
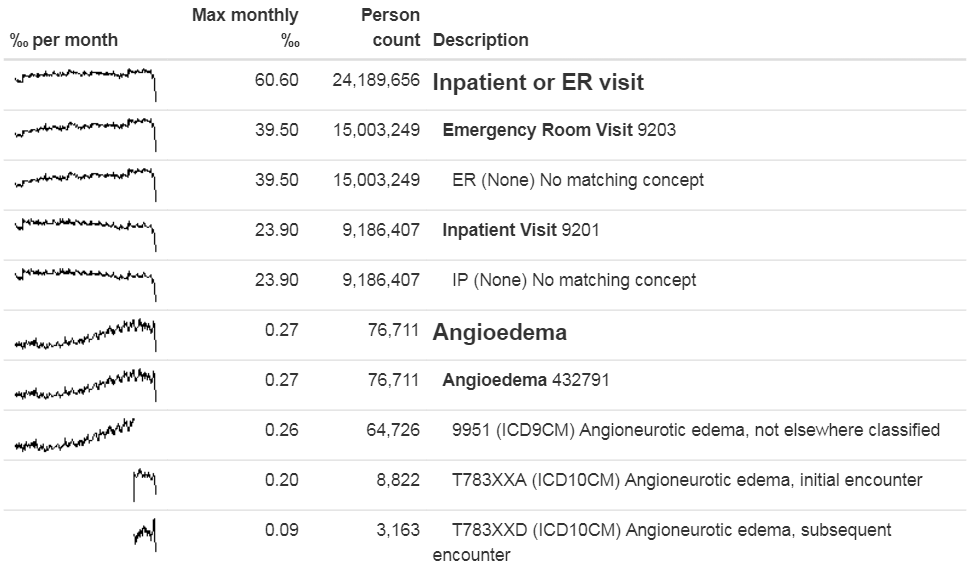
図 15.8: 血管性浮腫のコホート定義で使用されるソースコード
次に、標準コンセプトコードにマッピングされていない孤立したソースコードを検索できます。ここでは、標準コンセプト「Angioedema」を検索し、名前の一部として「Angioedema」または提供する同義語を含むコードやコンセプトを探します。
orphans <- findOrphanSourceCodes(connectionDetails,
cdmDatabaseSchema = cdmDbSchema,
conceptName = "Angioedema",
conceptSynonyms = c("Angioneurotic edema",
"Giant hives",
"Giant urticaria",
"Periodic edema"))
View(orphans)| コード | 説明 | 語彙ID | 全体のカウント |
|---|---|---|---|
| T78.3XXS | Angioneurotic edema, sequela(血管神経性浮腫、続発症) | ICD10CM | 508 |
| 10002425 | Angioedemas(血管性浮腫) | MedDRA | 0 |
| 148774 | Angioneurotic Edema of Larynx(喉頭の血管神経性浮腫) | CIEL | 0 |
| 402383003 | Idiopathic urticaria and/or angioedema(特発性蕁麻疹かつ/または血管性浮腫) | SNOMED | 0 |
| 232437009 | Angioneurotic edema of larynx(喉頭の血管神経性浮腫) | SNOMED | 0 |
| 10002472 | Angioneurotic edema, not elsewhere classified(血管神経性浮腫、他に分類されない) | MedDRA | 0 |
データで実際に使用されている潜在的なオーファンコード(上位層も下位層もない)として見つかったのは「Angioneurotic edema, sequela(血管神経性浮腫、続発症)」のみであり、これは血管性浮腫にマッピングすべきではありません。したがって、この分析では、欠落しているコードは発見されませんでした。
15.7 まとめ
ほとんどの観察医療データは研究のために収集されたものではありません。
データの品質チェックは研究に不可欠な要素です。データが研究目的に十分な品質かどうかを判断するには、データの品質を評価する必要があります。
一般に研究目的でデータの品質を評価すべきであり、特定の研究においては特に慎重に評価すべきです。
データ品質の一部は、Data Quality Dashboardのような大規模な事前に定義されたルールに基づいて自動的に評価することができます。
特定の研究に関連するコードのマッピングを評価するための他のツールも存在します。
15.8 演習
前提条件
これらの演習では、セクション 8.4.5で説明されているように、R、R-Studio、およびJavaがインストール済みであると想定します。また、SqlRender、DatabaseConnector、ACHILLES、およびEunomiaパッケージも必要です。これらは、以下のコマンドでインストールできます:
install.packages(c("SqlRender", "DatabaseConnector", "remotes"))
remotes::install_github("ohdsi/Achilles")
remotes::install_github("ohdsi/DataQualityDashboard")
remotes::install_github("ohdsi/Eunomia", ref = "v1.0.0")Eunomiaパッケージは、ローカルのRセッション内で実行されるCDMのシミュレーションデータセットを提供します。接続情報は以下のコマンドで取得できます:
CDMデータベーススキーマは「main」です。
演習 15.1 Eunomiaデータベースに対してACHILLESを実行してください。
演習 15.2 Eunomiaデータベースに対してData Quality Dashboardを実行してください。
演習 15.3 DQDのチェックリストを抽出してください。
解答例は付録 E.10を参照してください。