第 8 章 OHDSI 分析ツール
著者: Martijn Schuemie & Frank DeFalco
OHDSIは、患者レベルの観察データに対するさまざまなデータ分析のユースケースをサポートするための幅広いオープンソースツールを提供しています。これらのツールの共通点は、すべて共通データモデル(CDM)を使用して1つ以上のデータベースと相互にやりとりできることです。さらに、これらのツールはさまざまなユースケースに対して分析を標準化します。ゼロから始める必要はなく、標準テンプレートに入力することで分析を実装できます。これにより、分析が容易になり、再現性と透明性が向上します。例として、発生率を計算する方法は無数にあるように思われますが、OHDSIツールではいくつかの選択肢で指定でき、同じ選択肢を選んだ人は同じ方法で発生率を計算します。
本章では、最初に分析を実装するさまざまな方法を説明し、分析で採用できる戦略について説明します。次に、さまざまなOHDSIツールとそれらがどのようにさまざまなユースケースにどのように適合するかを検討します。
8.1 分析の実装
図 8.1 は、CDMを使用してデータベースに対して研究を実装するために選択できるさまざまな方法を示しています。
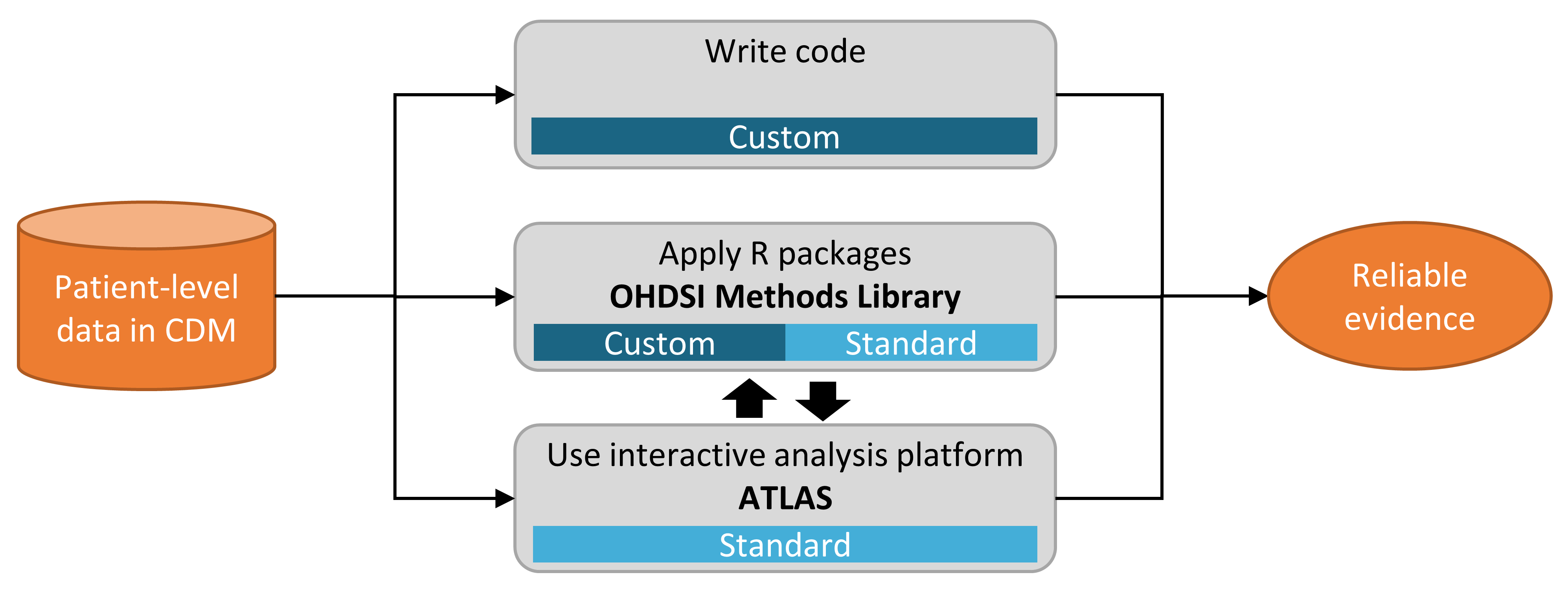
図 8.1: CDMのデータに対する分析を実装するさまざまな方法
研究を実装するための主なアプローチは3つあります。最初の方法は、OHDSIが提供するツールを一切使用しないカスタムコードを作成することです。R、SAS、またはその他の言語で新規の分析を作成することができます。これにより最大の柔軟性が得られ、特定の分析がツールでサポートされていない場合は唯一の選択肢となるかもしれません。しかし、この方法は高度な技術、時間、労力を必要とし、分析が複雑になるほどコードのエラーを避けることが難しくなります。
2番目の方法は、Rで分析を開発し、OHDSI Methods Libraryのパッケージを利用する方法です。少なくとも、SqlRenderとDatabaseConnectorのパッケージを使用できます。これらのパッケージについては、第9章で詳しく説明していますが、PostgreSQL、SQL Server、Oracleなどのさまざまなデータベースプラットフォーム上で同じコードを実行することができます。また、CohortMethodやPatientLevelPredictionなどのパッケージでは、CDMに対する高度な分析のためのR関数が提供されており、コードから呼び出すことができます。これには依然として高度な専門知識が必要ですが、検証済のMethods Libraryのコンポーネントを再利用することで、完全にカスタムコードを使用する場合よりも効率的に作業を進めることができ、エラーが発生する可能性も低くなります。
3番目の方法は、プログラマーでなくても幅広く分析を効率的に実行できるウェブベースのツール、ATLASを使用することです。ATLASはMethods Librariesを使用しますが、分析をデザインするための単純なグラフィカルインターフェイスを提供し、多くの場合、分析を実行するために必要なRコードを生成します。ただし、Methods Libraryで利用可能なすべてのオプションをサポートしているわけではありません。大半の研究はATLASを通じて行うことができると予想されますが、2番目の方法が提供する柔軟性を必要とする研究もあります。
ATLASとMethods Libraryは独立したものではありません。ATLASで呼び出される複雑な分析の一部は、Methods Libraryのパッケージへの呼び出しを通じて実行されます。同様に、Methods Libraryで使用されるコホートは、多くの場合、ATLASでデザインされています。
8.2 分析戦略
CDMに対する分析を実装するための戦略に加え、例えばカスタムコーディングやMethods Libraryで提供される標準分析コードの利用など、エビデンスを生成するための分析技術を使用する複数の戦略もあります。図 8.2 は、OHDSIで採用されている3つの戦略を示しています。
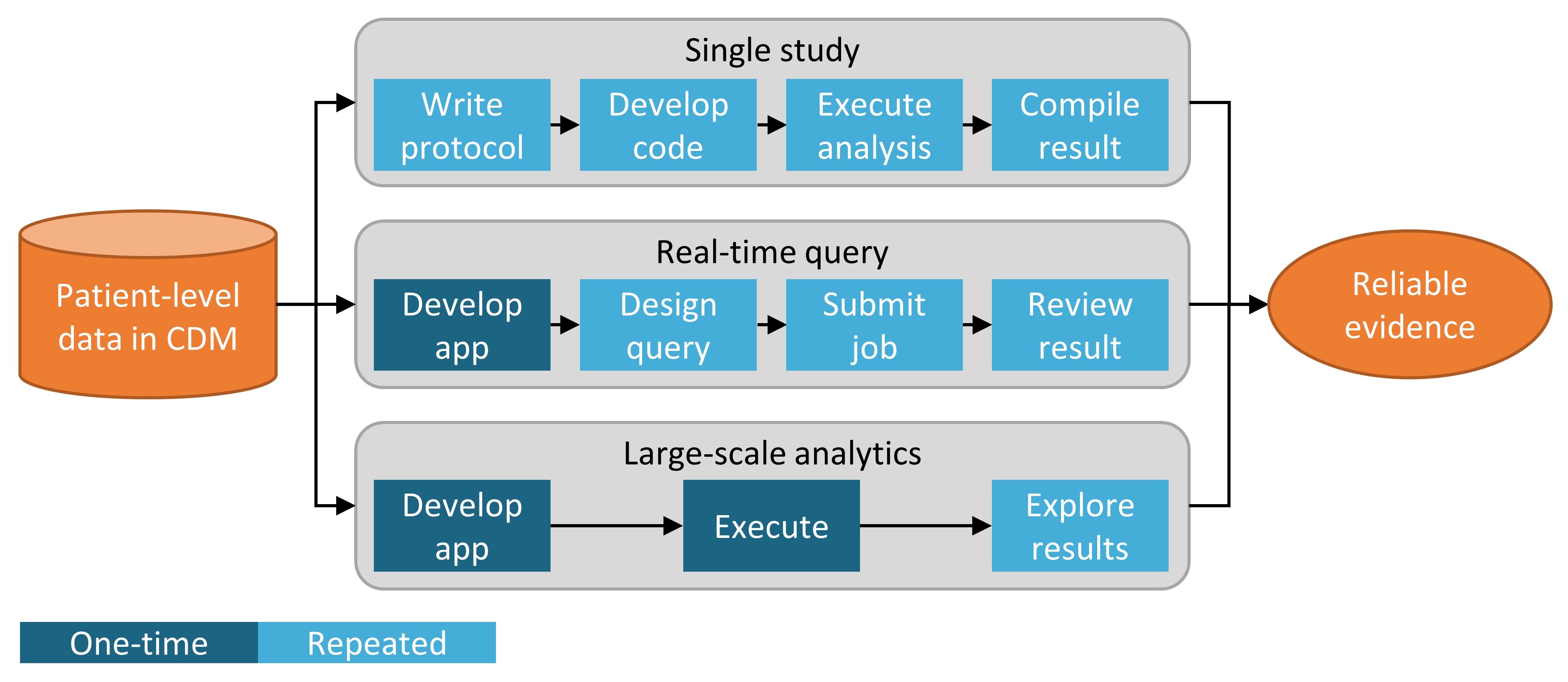
図 8.2: (臨床の)問いに対するエビデンスを生成するための戦略
最初の戦略では、各分析を個別の研究として扱います。分析はプロトコルで事前に規定し、コードとして実装し、データに対して実行し、その後、結果をまとめ解釈します。各課題(リサーチクエスチョン)ごとに、すべてのステップを繰り返す必要があります。このような分析の一例は、レベチラセタムとフェニトインを比較した際の血管性浮腫のリスクに関するOHDSI研究です(Duke et al. 2017)。 ここでは、まずプロトコルが作成され、OHDSI Methods Libraryを使用した分析コードが開発され、OHDSIネットワーク全体で実行され、結果がまとめられて学術誌に公表されました。
第二の戦略では、リアルタイムまたはほぼリアルタイムで特定のクラスの問いに答えられるアプリケーションを開発します。アプリケーションが開発されると、ユーザーはクエリをインタラクティブに定義し、それを送信して結果を表示できます。この戦略の一例は、ATLASのコホート定義および生成ツールです。このツールは、ユーザーが複雑さの異なるコホート定義を指定し、データベースに対してその定義を実行して、さまざまな適格基準と除外基準を満たす人数を確認することができます。
第三の戦略では、同様に問いに焦点を当てますが、その問いのクラス内のすべてのエビデンスを網羅的に生成しようとします。ユーザーは、さまざまなインターフェースを通じて必要に応じてエビデンスを探索できます。一例は、うつ病治療の効果に関するOHDSI研究です(Schuemie, Ryan, et al. 2018)。 この研究では、すべてのうつ病治療が、4つの大規模な観察研究データベースで関心のあるアウトカムの大規模なセットに対して比較されました。17,718の実証的にキャリブレーションされたハザード比と広範な研究診断を含む結果の全セットは、インタラクティブなウェブアプリ 39 で利用できます。
8.3 ATLAS
ATLAS は、OHDSI コミュニティが開発した、標準化された患者レベルの観察データを CDM 形式で分析する設計と実行を支援する、無料で公開されているウェブベースのツールです。ATLAS は、OHDSI WebAPI と組み合わせてウェブアプリケーションとして展開され、通常は Apache Tomcat 上でホストされます。リアルタイム分析を行うには、CDM 内の患者レベルデータへのアクセスが必要であるため、通常は組織のファイアウォールのバックにインストールされます。ただし、パブリックなATLAS40も存在し、このATLASインスタンスは少数の小規模なシミュレーションデータセットにしかアクセスできませんが、テストやトレーニングなど、多くの目的に使用できます。パブリックなATLASインスタンスを使用して効果の推定や予測研究を完全に定義し、研究を実行するためのRコードを自動的に生成することも可能です。そのコードは、ATLASとWebAPIをインストールすることなく、利用可能なCDMがインストールされている環境であればどこでも実行できます。
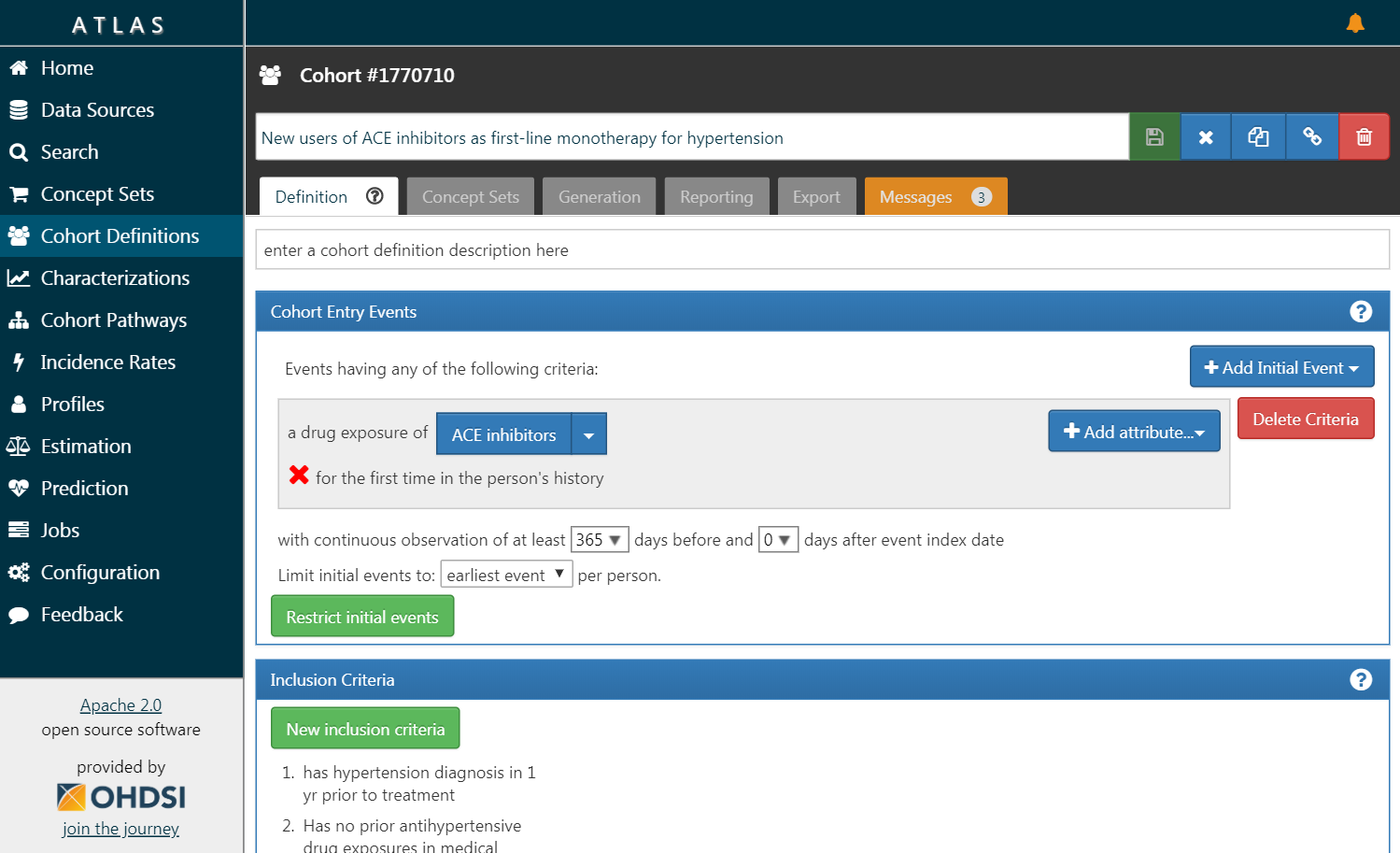
図 8.3: ATLASユーザインタフェース
図 8.3 にATLASのスクリーンショットを示します。左側にはATLASの様々な機能を示すナビゲーションバーがあります。
- データソース
-
データソースは、ATLASプラットフォームに構成された各データソースの記述的で標準化されたレポートをレビューする機能を提供します。この機能は大規模分析戦略を用いており、すべての記述は事前に計算されています。データソースについては、第11章で説明します。
- ボキャブラリ検索
-
ATLASはOMOP標準化ボキャブラリを検索し、これらのボキャブラリにどのようなコンセプトが存在するのか、そしてそのコンセプトをどう適用するかを理解するための機能を提供します。この機能については、第5章で議論されています。
- コンセプトセット
-
コンセプトセットは、標準化された分析全体で使用するコンセプトのセットを識別するために使用できる論理式のコレクションを作成する機能を提供します。コンセプトセットは単純なコードや値のリストよりも高度な機能を提供します。コンセプトセットは、標準化ボキャブラリからの複数のコンセプトと、関連するコンセプトの適格や除外を指定するための論理インジケータを組み合わせて構成されます。ボキャブラリの検索、コンセプトセットの特定、そしてコンセプトセットを決定するために使用する論理の指定は、分析プランで使用されることが多い難解な医療用語を定義するための強力なメカニズムとなります。これらのコンセプトセットはATLAS内に保存され、その後の分析の一部としてコホート定義や分析仕様に使用できます。
- コホート定義
-
コホート定義は、一定期間内に1つまたは複数の基準を満たす人のセットを構築する機能であり、これらのコホートはその後のすべての分析の入力の基礎として使用されます。この機能については、第10章で説明します。
- 特性評価
-
特性評価は、定義された1つまたは複数のコホートを調査し、これらの患者集団の特性を要約するための分析機能です。この機能はリアルタイムクエリ戦略を用いており、第11章で説明しています。
- コホート経路
-
コホート経路は、1つまたは複数の集団内で発生する臨床イベントのシーケンスを観察できる分析ツールです。この機能については、第11章で説明されています。
- 発生率
-
発生率は、対象集団内のアウトカムの発生率を推定するためのツールです。この機能については、第11章で説明されています。
- プロファイル
-
プロファイルは、個々の患者の縦断的観察データを調査し、特定の個人に起こっている状況を要約するためのツールです。この機能はリアルタイムクエリ戦略を使用します。
- 集団レベルの推定
-
推定は、比較コホートデザインを使用して集団レベルの効果推定研究を定義し、1つまたは複数の対象コホートと比較コホート間の比較の一連の結果を調査することができます。この機能はコーディングが不要で、リアルタイムクエリ戦略を実装していると言えます。この機能については第12章で説明されています。
- 患者レベルの予測
-
予測機能は機械学習アルゴリズムを適用して、患者レベルの予測分析を行い、特定のターゲット曝露内でアウトカムを予測することができます。この機能もリアルタイムクエリ戦略を実装しており、コーディングが不要です。第13章で説明されています。
- ジョブ
-
ジョブメニュー項目を選択して、WebAPIを通じて実行されているプロセスの状態を確認できます。ジョブは、コホートの生成やコホートの特性評価のレポートの計算など、長時間実行されるプロセスであることが多いです。
- 構成
-
構成メニュー項目を選択して、ソース構成セクションに構成されたデータソースを確認できます。
- フィードバック
-
フィードバックリンクをクリックすると、ATLASの課題ログにアクセスすることにより、新しいイシューのログの記録や既存のイシューの検索ができます。新しい機能や拡張機能のアイデアがある場合は、開発コミュニティに伝える場所としても利用できます。
8.3.1 セキュリティ
ATLASとWebAPIは、プラットフォーム全体で機能やデータソースへのアクセスを制御するための細かいセキュリティモデルを提供します。セキュリティシステムはApache Shiroライブラリを活用して構築されています。セキュリティシステムの詳細は、オンラインのWebAPIセキュリティWikiで確認できます 41。
8.3.2 ドキュメント
ATLASのドキュメントは、ATLAS GitHubリポジトリのWikiでオンラインで確認できます 42。 このWikiには、さまざまなアプリケーション機能に関する情報や、オンラインビデオチュートリアルへのリンクが含まれています。
8.4 Methods Library
OHDSI Methods libraryは、図 8.4 に示されているオープンソースのRパッケージのコレクションです。

図 8.4: OHDSI Methods Libraryのパッケージ
これらのパッケージは、CDM内のデータから始まり、推定値やそれを裏付ける統計、図表を生成する完全な観察研究を実施するためのR関数を提供しています。これらのパッケージはCDM内の観察データと直接やりとりし、第9章で説明されているような完全にカスタマイズされた分析にクロスプラットフォームの互換性を提供するために用いることも、集団特性の評価(第11章)、集団レベルの効果推定(第12章)、患者レベルの予測(第13章)のための高度な標準化分析を提供することもできます。Methods libraryは、透明性、再現性、異なるコンテキストでのメソッドの操作特性の測定値、そのメソッドによって生成される推定値やその後の実証的キャリブレーションなど、過去や現在の研究から学んだベストプラクティスをサポートしています。
Methods libraryはすでに多くの公表された臨床研究 (Boland et al. 2017; Duke et al. 2017; Ramcharran et al. 2017; Weinstein et al. 2017; Wang et al. 2017; Ryan et al. 2017, 2018; Vashisht et al. 2018; Yuan et al. 2018; Johnston et al. 2019) で使用されており、方法論の研究にも利用されています (Schuemie et al. 2014, 2016; Reps et al. 2018; Tian, Schuemie, and Suchard 2018; Schuemie, Hripcsak, et al. 2018; Schuemie, Ryan, et al. 2018; Reps, Rijnbeek, and Ryan 2019)。Methods library内のメソッドの実装の妥当性については第17章で説明されています。
8.4.1 大規模分析サポート
すべてのパッケージで組み込まれている重要な機能の一つは、多くの分析を効率的に実行できることです。例えば、集団レベルの推定を行う場合、CohortMethodパッケージは多数の曝露とアウトカムに対して効果量の推定を行うことを可能にし、さまざまな分析設定を使用して、必要な中間データセットや最終データセットを計算するための最適な方法を自動的に選択します。共変量の抽出や、一つのターゲット・比較対照ペアに対して複数のアウトカムで使用される傾向スコアモデルの適合など、再利用可能なステップは一度だけ実行されます。可能な場合は、計算リソースを最大限に活用するために計算は並列処理されます。
この計算効率により、大規模な分析が可能になり、多くの課題に一度に回答することができます。また、コントロール仮説(ネガティブコントロールなど)を含めることで、当社の手法の運用特性を測定し、第18章 で説明されているように、実証的なキャリブレーションを行うことも不可欠です。
8.4.2 ビッグデータ対応
Methods Libraryは、非常に大規模なデータベースに対しても大量のデータを含む計算を実行できるようにデザインされています。これは次の三つの方法で実現されます:
- 大部分のデータ操作はデータベースサーバー上で実行されます。分析は通常、データベース内の全データのごく一部しか必要としないため、Methods LibraryはSqlRenderやDatabaseConnectorパッケージを介して関連データの前処理や抽出をする高度な操作をサーバー上で実行できるようにします。
- 大量のローカルデータオブジェクトはメモリ効率の良い方法で保存されます。ローカルマシンにダウンロードされるデータについては、Methods Libraryはffパッケージを使用して大規模データオブジェクトを保存、処理します。これにより、メモリに収まらないほど大きなデータでも処理することが可能です。
- 必要に応じて高性能コンピューティングが適用されます。例えば、Cyclopsパッケージは、Methods Library全体で使用される非常に効率的な回帰エンジンを実装しており、これにより通常は適合できない大規模な回帰(多くの変数、大量の観測値)を実行することができます。
8.4.3 ドキュメント
Rはパッケージを文書化するための標準的な方法を提供しています。各パッケージには、パッケージに含まれるすべての関数とデータセットを文書化したパッケージマニュアルがあります。すべてのパッケージマニュアルは、Methods Libraryのウェブサイト45、パッケージのGitHubリポジトリ、CRANで利用できます。さらに、Rの内部からパッケージマニュアルを参照するにはクエスチョンマークを使用します。例えばDatabaseConnectorパッケージを読み込んだ後、コマンド?connectを入力すると「connect」関数に関するドキュメントが表示されます。
パッケージマニュアルに加えて、多くのパッケージはビネットが提供されています。ビネットは、特定のタスクを実行するためにパッケージをどのように使用するかを説明した詳細なドキュメントです。例えば、一つのビネット46では、CohortMethodパッケージを使用して複数の分析を効率的に実行する方法が説明されています。ビネットはMethods Libraryのウェブサイト、パッケージのGitHubリポジトリ、CRANで入手可能なパッケージはCRANでも見つけることができます。
8.4.4 システム要件
システム要件を検討する際には、二つのコンピューティング環境が関連してきます:データベースサーバーと分析ワークステーションです。
データベースサーバーはCDM形式の観察医療データを保持する必要があります。Methods Libraryは、従来のデータベースシステム(PostgreSQL、Microsoft SQL Server、Oracle)、パラレルデータウェアハウス(Microsoft APS、IBM Netezza、Amazon Redshift)、に加えビッグデータプラットフォーム(Impala経由でのHadoop、Google BigQuery)など、幅広いデータベース管理システムをサポートしています。
分析ワークステーションは、Methods Libraryがインストールされ実行される場所です。これがローカルマシン(例えば、ノートパソコン)か、RStudio Serverが実行されるリモートサーバーかに関わらず、Rがインストールされている必要があります。可能であればRStudioも一緒にインストールすることをお勧めします。また、Methods LibraryではJavaがインストールされている必要があります。分析ワークステーションはデータベースサーバーに接続できる必要があり、具体的には、両者の間にファイアウォールがある場合は、ワークステーションでデータベースサーバーのアクセスポートが開いている必要があります。一部の分析は計算集中的であるため、複数のプロセッサコアと十分なメモリを持つことが分析の高速化につながります。少なくとも4コアと16ギガバイトのメモリを推奨します。
8.4.5 インストール方法
OHDSI Rパッケージを実行するために必要な環境をインストールするための手順は次の通りです。インストールする必要があるものは4つあります:
- Rは統計的コンピューティング環境です。基本的なユーザインターフェースとして主にコマンドラインインターフェースを提供します。
- Rtoolsは、WindowsでRパッケージをソースからビルドする際に必要なプログラム一式です。
- RStudioは、Rを使いやすくするIDE(統合開発環境)です。コードエディタ、デバッグ、およびビジュアルツールが含まれています。素晴らしいR体験を得るために、これを使用ください。
- Javaは、OHDSI Rパッケージの一部のコンポーネント、例えばデータベースへの接続に必要なコンポーネントを実行するために必要なコンピューティング環境です。
以下では、Windows環境でのそれぞれのインストール方法を説明します。
Rのインストール
- https://cran.r-project.org/ で、図 8.5 に示されるように「Download R for Windows 」、「base」 の順にクリックし、ダウンロードしてください。
図 8.5: CRANからのRのダウンロード
- ダウンロードが完了したら、インストーラを実行します。2つの例外を除いて、すべてデフォルトのオプションを使用してください: まず、プログラムファイルにはインストールしない方が良いでしょう。代わりに、図 8.6 のように、CドライブのサブフォルダとしてRを作成します。次に、RとJavaのアーキテクチャの違いによる問題を回避するため、図 8.7 のように32ビットアーキテクチャを無効にします。
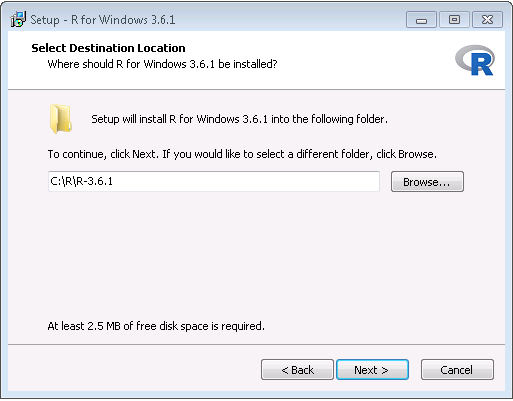
図 8.6: Rフォルダの設定
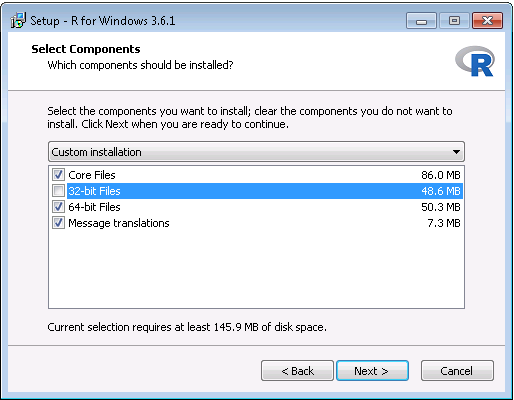
図 8.7: 32ビットバージョンのRを無効化
完了すると、スタートメニューからRを選択できるようになります。
Rtoolsのインストール
https://cran.r-project.org/ にアクセスし、「Download R for Windows」をクリックし、次に「Rtools」をクリックして、最新版のRtoolsをダウンロードします。
ダウンロードが完了後、インストーラを実行します。すべてデフォルトのオプションを選択します。
RStudioのインストール
- https://www.rstudio.com/ にアクセスし、「Download RStudio」またはRStudioの下の「ダウンロード」ボタンをクリックし、無料版を選択し、図 8.8 に示されるようにWindows用のインストーラをダウンロードします。
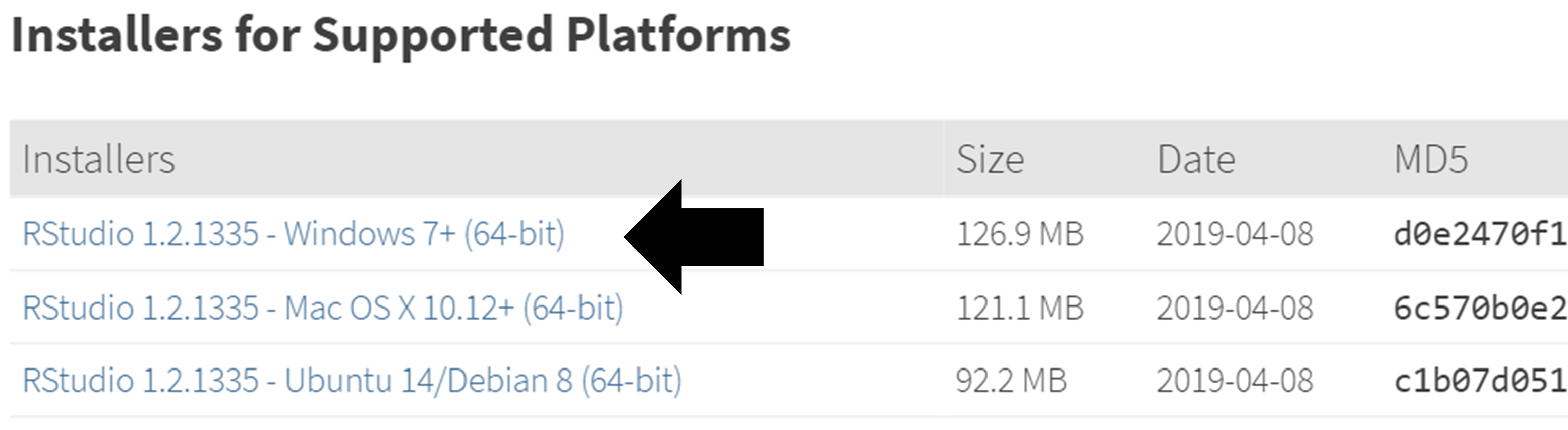
図 8.8: RStudioのダウンロード
- ダウンロードが完了後、インストーラを実行します。すべてデフォルトのオプションを選択してください。
Javaのインストール
- https://java.com/en/download/manual.jsp にアクセスし、図 8.9 に示されるように、Wiindows64ビット版のインストーラを選択します。32ビット版のRもインストールしている場合には、Javaも32ビット版をインストールしなければなりません。

図 8.9: Javaのダウンロード
- ダウンロード後、インストーラを実行してます。
インストールの確認
これで準備は整ったはずですが、念のため確認しておきましょう。Rを起動し、下記のようにタイプしてください。
install.packages("SqlRender")
library(SqlRender)
translate("SELECT TOP 10 * FROM person;", "postgresql")## [1] "SELECT * FROM person LIMIT 10;"
## attr(,"sqlDialect")
## [1] "postgresql"この関数はJavaを使用するので、すべてがうまくいけば、RとJavaの両方が正しくインストールされていることがわかります!
もう一つのテストは、ソースパッケージがビルドできるかどうかを確認することです。以下のRコードを実行して、OHDSI GitHubリポジトリからCohortMethodパッケージをインストールします:
8.5 展開戦略
ATLASやMethods Libraryを含むOHDSIツールスタック全体を組織内で展開することは、非常に困難な作業です。依存関係がある多くのコンポーネントを考慮し、設定を行う必要があります。このため、二つの取り組みが、スタック全体を一つのパッケージとしてインストールできる統合展開戦略を開発しました。一部の仮想化技術を使用して、これを実現します。それは、BroadseaおよびAmazon Web Services(AWS)です。
8.5.1 Broadsea
Broadsea47はDockerコンテナ技術48を使用しています。OHDSIツールは依存関係とともに、Dockerイメージと呼ばれる単一のポータブルなバイナリファイルにパッケージ化されています。このイメージはDockerエンジンサービス上で実行でき、すべてのソフトウェアがインストールされるとすぐに実行可能な仮想マシンが作成されます。DockerエンジンはMicrosoft Windows、MacOS、Linuxなどのほとんどのオペレーティングシステムで利用可能です。Broadsea Dockerイメージには、Methods LibraryやATLASを含む主なOHDSIツールが含まれています。
8.5.2 Amazon AWS
Amazonは、AWSクラウドコンピューティング環境でボタンをクリックするだけでインスタンス化できる二つの環境を用意しています:OHDSI-in-a-Box49とOHDSIonAWS50です。
OHDSI-in-a-Boxは特に学習環境として作成さたものであり、OHDSIコミュニティが提供するほとんどのチュートリアルで使用されています。これには多くのOHDSIツール、サンプルデータセット、RStudio、その他のサポートソフトウェアが低コストの単一のWindows仮想マシンに含まれています。PostgreSQLデータベースは、CDMの保存と、ATLASからの中間結果の保存の両方に使用されます。OMOP CDMデータマッピングとETLツールも、OHDSI-in-a-Boxに含まれています。OHDSI-in-a-Boxのアーキテクチャは、図 8.10 に示されています。
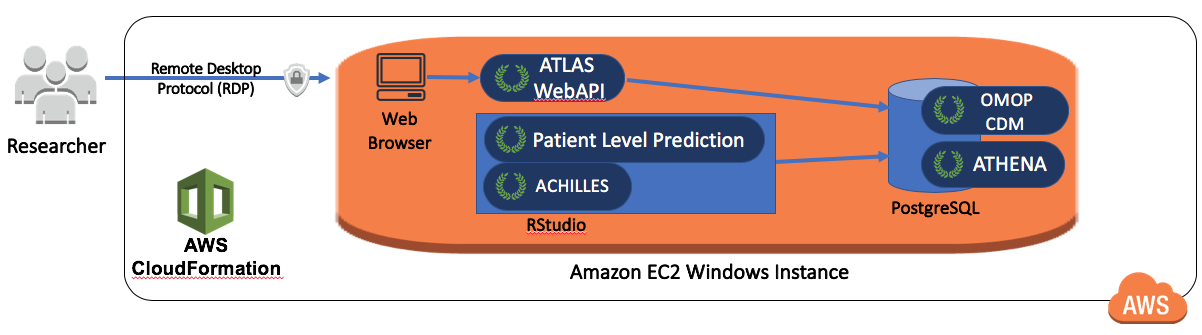
図 8.10: OHDSI-in-a-BoxのAmazon Web Servicesアーキテクチャ
OHDSIonAWSは、企業向け、マルチユーザー対応、拡張性や耐障害性に優れたOHDSI環境のためのリファレンスアーキテクチャであり、組織がデータ分析を行う際に使用することができます。 複数のサンプルデータセットが含まれており、組織の実際のヘルスケアデータを自動的にロードすることも可能です。 データはAmazon Redshiftデータベースプラットフォームに配置され、OHDSIツールによってサポートされます。 ATLASの中間結果はPostgreSQLデータベースに保存されます。ユーザーはフロントエンドで、ウェブインターフェース(RStudio Serverを活用)を通じてATLASやRStudioにアクセスできます。RStudioにはOHDSI Methods Libraryがすでにインストールされており、データベースへの接続に使用できます。OHDSIonAWSの自動展開はオープンソースであり、組織の管理ツールやベストプラクティスを含めるようにカスタマイズできます。OHDSIonAWSのアーキテクチャは図 8.11 に示されています。
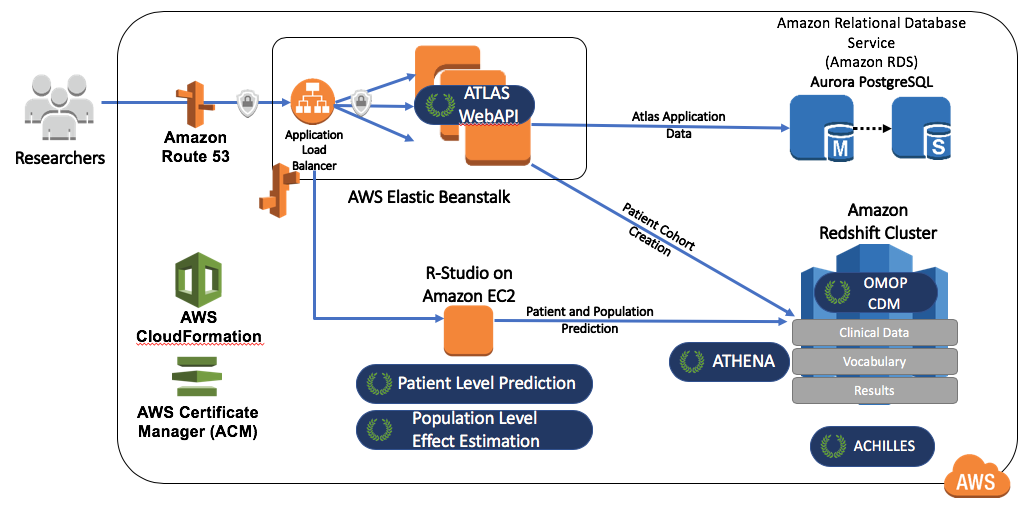
図 8.11: OHDSIonAWSのAmazon Web Servicesアーキテクチャ